زیست شناسی سامانه ای چیست؟ – آموزش بیولوژی سیستم به زبان ساده
بالا بردن دست یکی از سادهترین حرکات انسان است. اما برای انجام این حرکت ساده چندین واکنش مولکولی برای سنتز انتقالدهندههای عصبی، انتقال آنها به پایانههای آکسون، انتقال جریان بین نورونها، انتقال جریان از نورون به ماهیچه، انقباض ماهیچه، هماهنگی دست با چشم و هماهنگی دست با مغز انجام میشود. درک رابطه و واکنشهای بین مولکولهای زیستی به پیشبینی تغییر سیستمهای زیستی در شرایط بیماری یا سلامت و برهمکنش با مولکولهای دارو کمک فراوانی میکند. اما درک این روابط بسیار پیچیده بدون استفاده از معادلات ریاضی کار دشوار و زمانبری است. زیست شناسی سامانه ای با ادغام زیستشناسی، تکنولوژی و روابط محاسباتی امکان در درک این روابط و پیشبینی تغییرات آن را فراهم میکند. در این مطلب از مجله فرادرس روشهای مدلسازی زیستشناسی سامانهای را همراه چند نرمافزار کاربردی توضیح میدهیم.
زیست شناسی سامانه ای چیست ؟
پیشبینی کمی میزان گرمای ایجاد شده در پایان یک واکنشهای شیمیایی و یا نیروی لازم برای بالا جسم ده کیلویی کار دشواری نیست. اما در سیستمهای زیستی هزاران واکنش و مسیر بیوشیمایی همزمان با هم بدن موجود تغییر ایجاد میکنند و پیشبینی مقدار عددی این واکنشهای کار بسیار دشوار و زمانبری است.
«زیست شناسی سامانه ای» (Systems Biology) روشی در زیستشناسی و پزشکی است که از تکنولوژی، محاسبات ریاضی، فیزیک و مدلسازی برای فهم ساختار و عملکرد سیستم موجود زنده استفاده میکند. به کمک این روش میتوان فهمید اجزای سیستم زیستی کدامند. چه ارتباط با هم دارند و از ارتباط آنها چه نتیجهای ایجاد میشود. به علاوه میتوان با استفاده از ریاضی و فیزیک ارتباط عددی این فرایندها را محاسبه کرد. برای مثال به کمک مدلهای کامپیوتری این روش میتوان زمان اثر دارو پس تجویز، میزان «بههمچسبیدگی مولکولهای دارو» (Aggregation) در خون، pH مناسب برای اثر دارو در بافت هدف و اثر تغییرات دما بر ساختار دارو را پیشبینی کرد. در نتیجه هزینه کمتری صرف هزینه دارو خواهد شد و عوارض جانبی آن در بدن کاهش مییابد.
در زیست شناسی سامانه ای برخلاف روشهای قبلی موجود زنده سیستمی یکپارچه در نظر گرفته میشود. برای مثال در پروژه ژنوم انسان از رویکرد زیست شناسی سامانه ای استفاده شده است. در این پروژه تمام توالی، عملکرد و ارتباط بین عملکرد تمام ژنهای انسان، در تمام کروموزومهای اتوزوم و جنسی و در تمام سلولها بررسی شد.
اطلاعات طراحی مدل
برای طراحی معادلات ریاضی و شبیهسازی فرایندهای زیستشناسی به اطلاعات اولیه در مورد ساختار مولکولهای شرکتکننده و روابط بین آنها نیاز است. این اطلاعات را میتوان به وسیله روشهای آزمایشگاهی کلاسیک، روشهای مولکولی جدید، بررسی پژوهشهای قبلی یا پایگاه دادههای بیوانفورماتیک به دست آورد. بیوانفورماتیک روشی برای طبقهبندی، ذخیره و آنالیز اطلاعات زیستشناسی در سیستمهای دیجیتالی است. متخصصین این رشته اطلاعات ژنومیک، پروتئومیک، ترانسکریپتومیک و متابولومیک را طبقهبندی و نرمافزارهای بهروز برای آنالیز اطلاعات طراحی میکنند.
- در ژنومیک توالی، عملکرد، ارتباط بین ژنها و ارتباط ژنها با رشد موجود زنده را بررسی میکند. اطلاعات طبقهبندی شده در پایگاه دادههای ژنومیک برای مدلسازی مسیرهای تنظیم بیان ژن و پیشبینی اثر تغییرات ژنتیکی بر سلامت موجود زنده استفاده میشود. Gene Bank یکی از پایگاه دادههای ذخیره توالی ژنی است. در این پایگاه علاوه بر گونه انسان اطلاعات ژنتیکی بسیاری از میکرواورگانیسمها ازجمله باکتری ای. کلی ذخیره شده است. در ترانسکریپتومیک توالی، ساختار و عملکرد تمام RNAهای موجود (tRNA، mRNA، rRNA، iRNA) زنده بررسی میشود.
- در پروتئومیک تمام پروتئینهای ساختاری و عملکردی یک موجود زنده، انداتم یا سلول بررسی میشود. زمان و جایگاه سلولی بیان پروتئینها، سرعت سنتز، تجزیه و حالت پایدار پروتئین، مکانیسم تغییر پروتئینها (برای مثال اضافه شدن متیل پس از ترجمه)، انتقال پروتئین بین اندامکهای سلولی، نقش پروتئین در مسیرهای متابولیسمی و برهمکنش پروتئینها با هم ویژگیهایی است که در پروتئومیک بررسی میشود. Protein Data Bank یکی از پایگاه دادههای پروتئومیک است. در این پایگاه داده توالی آمینواسید پروتئین، ساختار سهبعدی، عملکرد پروتئین و برهمکنش پروئین با پروتئینهای دیگر یا دارو ثبت شده است.
- متابولومیک تمام متابولیتهای سلول، مایعات و بافتهای بدن که در واکنشهای بیوشیمیایی شرکت میکنند یا در نتیجه این واکنشهات تولید میشوند را بررسی میکند. The Human Metabolome Database یکی از پایگاه دادههای در دسترس متابولومیک برای طبقهبندی ویژگیهای شیمیایی، بالینی و مولکولی متابولیتهای متابولیتهای کوچک محلول در چربی و آب بدن انسان است.
مدلسازی در زیست شناسی سامانه ای
مدل نمونهای انتزاعی و غیرواقعی اما در دسترس از یک فرایند یا مولکول است که ویژگیهای اصلی آن را نشان میدهد. برای مثال ممکن است در هر آزمایشگاهی امکان استخراج DNA وجود نداشته باشد، اما میتوان از دیتابیسهای DNA در هر آزمایشگاهی استفاده کرد.
در مدلهای زیست شناسی سامانه ای از قوانین فیزیکی برای بررسی واکنشهای شیمیایی با در نظر گرفتن ویژگیهای بیوشیمیایی و زیستی واکنشها استفاده میشود. برای مثال در مدلسازی واکنشهای آنزیمی از قوانین ترمودینامیک بهره برده میشود. در نتیجه یک نمونه در سیستم تعریف میشود و سیستم بر اساس فرمولهای ریاضی، تغییرات احتمالی و رفتار نمونههای مشابه در سیستم بدن موجود زنده را پیشبینی میکند.
در زیستشناسی آزمایشگاهی مدل موجود زندهای است که میتوان به کمک آن تغییر احتمالی یک فرایند زیستی را با روشهای آزمایشگاهی (روشهای تشخیص مولکولی، اسپکتروفوتومتری، رنگسنجی، الکترفورز و PCR) پیشبینی کرد. برای مثال باکتری ای. کلی و مخمر ساکارامیسز از مدلهای مناسب برای بررسی جهشهای ژنتیکی هستند. اختلاف زیاد نتایج پیشبینی شده بهوسیله مدل و روشهای آزمایشگاهی نشاندهنده مدلسازی اشتباه یا سادهسازی زیاد مدل است.
مراحل مدلسازی
شکل زیر مراحل مدلسازیهای زیست شناسی سامانه ای نشان داده شده است. در مرحله اول پروتئین یا واکنش بیوشیمیایی در سیستم زنده بررسی میشود. سیستم زنده در شکل باکتریهای ای. کلی را نشان میدهد که پروتئینهای آن بهوسیله رنگهای فلوئورسنس نشانهگذاری شده و زیر میکروسکوپ فلورسانس مشاهده میشود. «مدل ذهنی» (Mental Model) یکی از سادهترین روشهای مدلسازی است. در این روش تصور میشود سلول از دو آنزیم X (قرمز) و Y (آبی) تشکیل شده است و مولکولها بهوسیله انتشار در سلول پخش میشوند. در این مدل ساختارهای دیگر سلول برای سادهسازی حذف میشوند.
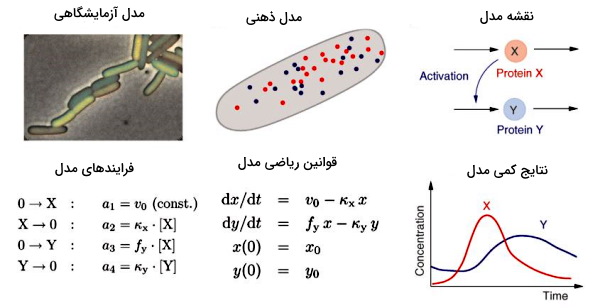
ترسیم «نقشه مدل» (Model Scheme) مرحله بعدی مدلسازی زیست شناسی سامانه ای است. در این روش میتوان با فلشها و نقطههای ساده سنتز یا تجزیه پروتئینهای مختلف را مشخص کرد. در مدل شکل بالا سنتز پروتئین X سنتز پروتئین Y را افزایش میدهد. در مرحله تمام فرایندهای انجام شده و سرعت انجام فرایندهای (دفعات انجام فرایند در واحد زمان) تعیین میشود. برای رسیدن به روابط ریاضی این مرحله واکنشهای زیستی، فرایندهای بیوشیمایی جدا از هم و ساده در نظر گرفته میشوند. در مرحله بعد روابط ریاضی فرایندها به معادلات ترمودینامیکی تبدیل میشود. در مثال بالا قوانین ترمودینامیک برای رابطه سرعت واکنش و غلظت پروتئینها در نظر گرفته شده است. در مرحله آخر با حل معادلات ریاضی میتوان نتیجه تغییرات واکنش را بررسی کرد. برای مثال در تصویر بالا میتوان رابطه تغییر زمان و غلظت رو پروتئین را پییشبینی کرد.
ویژگی های مدل چیست؟
هدف، معادلهها، حالت سیستم، متغیرها، ثابتها و پارامترها، رفتار مدل، فرایندها و حالت پایدار مدل ویژگیهایی است که در مدلسازی زیست شناسی سامانه ای باید به آنها توجه کرد.
- هدف: برای سنتز یک مولکول مشخص، همزمان تعداد زیادی فرایند در بدن انجام میشود. در مدلهای زیست شناسی سامانه ای برای سادهسازی معادلات ریاضی تغییرات یکی از پارامترهای سیستم برای مثال تغییرات غلظت در نظر گرفته میشود.
- معادلهها: هر مدل از معادلاتی تشکیل شده است که وضعیت عناصر اصلی سیستم در زمانهای مختلف فرایند را پیشبینی میکند. برای مثال معادلات مدلسازی سینتیکی رابطه ریاضی برقراری تعادل بین غلظت ترکیبات در طول واکنش نشان داده میشود.
- حالت سیستم: حالت سیستم برشی از سیستم در یک زمان مشخص است که بهوسیله متغیرها تعریف میشود. حالت سیستم در مدلهای قطعی و تصادفی تعریف متفاوتی دارد.
- سیستم قطعی: در مدلهای قطعی و کمی برای پیشبینی حالت سیستم در آینده باید اطلاعات کافی از تمام عناصر داشته باشیم. برای مثال در مدلهای سینتیکی، حالت سیستم غلظت تمام عناصر واکنش است و بر اساس تغییرات غلظت میتوان روند پیشرفت واکنش را پیشبینی کرد. در این مدلها حالت آینده سیستم به حالت این لحظه سیتم بستگی دارد.
- مدل تصادفی: در مدلهای تصادفی و کیفی حالت سیستم دامنه احتمال و عدد مولکولها حالت سیستم را تعیین میکند. برای مثال در «مدل بولن» (Boolean Model) برای تنظیم ژن عدد 0 نشاندهنده حذف ژن یا ژن غرفعال و عدد ۱ نشاندهنده وجود زن یا ژن فعال است. در این سیستمها چندین حالت با احتمالهای متفاوت برای آینده پیشبینی میشود.
- متغیر، ثابت و پارامتر: مقادیر عددی هر مدل در سه گروه متغیر، ثابت و پارامتر قرار میگردد. ثابتها مقادیر عددی مشخصی هستند که با قوانین ریاضی تعیین شدهاند. برای مثال ثابت گازها در معادلات ترمودینامیکی، Km آنزیمها و عدد آووگادو برای محاسبه وزن مولکولی از ثابتهایی هستند که در مدلسازی استفاده میشوند. متغیرها مقادیر عددی هستند که در طول فرایند تغییر میکنند. برای مثال در معادله $$PV= nRT$$، R ثابت گازهای ایدهآل (۸٫۳۱۴ J/mol.k) و P (فشار)، T (دما)، V (حجم) و n (مول) متغیرهای معادله هستند. پارامتر معمولا مقدار ثابتی است که با تغییر آن رفتار سیستم تغییر میکند و بر اساس مدل تعریف میشود. برای مثال غلظت آنزیم در مدلهای سینتیکی مقدار ثابت و پارامتر است، اما در بیان ژن یا تجزیه پروتئین تغییر میکند.
- رفتار مدل: رفتار یا «خروجی سیستم» (Output) بهوسیله اثر محیط بر سیستم (Input) و فرایندهای داخلی آن تعیین میشود. ارتباط بین اجزای ساختار یک سیستم (پارامتر، متغیر و ثابت) پاسخ سیستم (رفتار) به نیروهای داخلی و خارجی را نشان میدهد. به همین دلیل در زیست شناسی سامانهای سیستمهای با ساختار متفاوت ممکن است رفتار مشابهی داشته باشند.
- فرایندها: از فرایندهای ساختاری یا کیفی، مطلق یا وابسته (Deterministic Model)، مجزا یا گسسته (Discrete Model)، برگشتپذیر و تناوبی در مدلسازی سیستمهای زیستی استفاده میشود.
- کیفی: این فرایندها ارتباط بین اجزای مدل را نشان میدهند.
- مطلق: در این فرایندها حالت فرایند در هر لحظه را میتوان با استفاده از روابط ریاضی و مقادیر عددی پیشبینی کرد.
- گسسته: در این فرایندها پارامترهای مجزا در معادلات وارد میشوند.
- برگشتپذیر: در فرایندهای برگشتپذیر حالت تعادلی بین عناصر یک سیستم وجود دارد. در این فرایندها ورودی و خروجی سیستم به هم تبدیل میشوند. اما در فرایندهای برگشتناپذیر فرایند یکطرفه است. برای مثال تشکیل کمپلکس آنزیم-سوبسترا یک فرایند زیستی برگشتپذیر و تشکیل محصول از سوبسترا یک فرایند برگشتناپذیر است.
- تناوبی: در فرایندهای تناوبی سیستم مجموعهای از حالتها در نظر گرفته میشود.
- حالت پایدار: سیستمهای زیستی از فرایندهای بسیار پویایی تشکیل شده است که در بازه زمانی نانوثانیه انجام میشوند. در نظر گرفتن تمام این فرایندها مجموعهای از معادلات پیچیده ریاضی در اختیار فرد قرار میدهد که پیدا کردن رابطه بین عناصر زیستی را به فرایندی بسیار دشوار و زمانبر تبدیل میکند. به همین دلیل برای مدلسازی بهینه حالت پایدار فرایند زیستی در نظر گرفته میشود. حالت پایدار برشی از این فرایندها است که تمام پارامترها در بازه زمانی بسیار کوتاه مقدار ثابتی است.
روش های مدلسازی زیست شناسی سامانه ای
برای انتخاب بهترین روش مدلسازی سیستمهای زیستی لازم است پاسخ چند سوال را بدانیم. این مدل چه سوالی را پاسخ میدهد. این مدل برای براری ارتباط بین مشاهدات مختلف و اطلاعات قبلی طراحی میشود؟ مدل برای پیشبینی تغییر فرایند یا بررسی فرایندهای قبلی طراحی میشود؟ پس از پاسخ دادن به این پرسشها میتوانیم مدل ریاضی بهینه برای هدفمان را انتخاب کنیم. مدلهای شبکهای، مدلهای دستوری و مدلهای آماری سه روش محاسباتی در مدلسازیهای زیست شناسی سامانه ای هستند.
مدل شبکه ای زیست شناسی سامانه ای چیست ؟
شبکه نموداری است که بهوسیله نقطه یا «گره» (Node) و یال یا «لبه» (Edge) ارتباط بین اجزای سیستم را نشان میدهد. گرههای این مدل، مولکولهای زیستی یا ژنها و خطها برهمکنش یا ارتباط بین این مولکولها را نشان میدهد. این مدلها ویژگیهای ساختاری فرایندهای بیوشیمیایی را نشان میدهند. به کمک این مدل میتوان ویژگیها، حالت و برهمکنش صدها مولکول در یک فرایند زیستی پیچیده توضیح داد. «معادلات دیفرانسیل معمولی» (Ordinary Differential Equations | ODE)، «مدلهای تصادفی» (Stochastic Description)، مدل بولن و «مدل شبکه پتری» (Petri Net Models) مدلهای شبکهای متداولی هستند که در زیست شناسی سامانهای کاربرد دارند.
معادلات دیفرانسیل معمولی
به کمک این مدل میتوان رفتارهای پویا و ایستای واکنشهای شیمیایی را مدلسازی کرد. در این مدل از متغیرهای پیوسته (معمولا غلظت) در زمان پیوسته برای نشان دادن تغییرات حالت مطلق استفاده میشود.
برای مثال بخشی از واکنشهای متابولیکی گلیکولیز را بهوسیله این سیستم مدلسازی میکنیم. گلیکولیز یکی از واکنشهای متابولیکی اصلی در بدن است که مولکول گلوکز را به پیروات و ATP تبدیل میکند. شکل زیر واکنشهای ابتدایی گلیکولیز را نشان میدهد. که در آن S0 گلوکز خارج سلولی، S1 گلوکز سیتوپلاسم، S2 گلوکز ۶-فسفات (G6P)، S3 مسیرهای سنتزی دیگر، S4 فروکتوز ۱و ۶-بیس فسفات (F16P2)، A3 مولکول ATP و A2 مولکول ADP است.
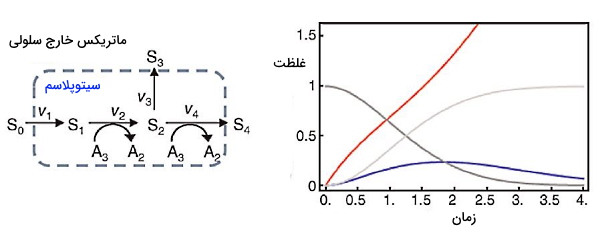
سرعت تبدیل ترکیبات این فرایند (دینامیک شبکه) را میتوان با معادلات دیفرانسیلی زیر نشان داد.
$$\frac{\text{d}S_1}{\text{d}t} = v_1 - v_2$$
$$\frac{\text{d}S_2}{\text{d}t} = v_2 - v_3 - v_4$$
$$\frac{\text{d}A_2}{\text{d}t} = - \frac{\text{d}A3}{\text{d}t}= v_2 + v_4$$
سرعت واکنشهای بیوشیمیایی را میتوان با استفاده از قوانین سینتیکی محاسبه کرد. بر این اساس سرعت واکنش از حاصلضرب غلظت سوبسترا و ثابت سرعت به دست میآید.
$$v_1=k_1$$ ،
$$v_2=k_2\times S_1\times A_3$$
$$v_3=k_3\times S_3$$
$$v_4=k_4\times S_2\times A_3$$
برای شبیهسازی تغییرات شبکه غلظت اولیه ATP را یک، غلظت اولیه ADP و کربوهیدراتهای داخل سلولی (S1 و S2) را صفر در نظر میمیگیریم. در این واکنش ATP مصرف شده و ADP تولید میشود. S1 بدون محدودیت و از ورود S0 به سلول تولید میشود، اما تولید S2 وابسته به ATP است. از آنجایی که S0 به شکل نامحدود وجود دارد، سیستم به حالت پایدار یا تعادل بیوشیمایی نمیرسد و غلظت ترکیبات در هر لحظه تغییر میکند. اما اگر غلظت ATP و ADP بهوسیله فرایندهای سلولی دیگر ثابت نگه داشته شود، سیستم به حالت پایدار میرسد. در این حالت سرعت تولید S1 برابر با S2 و صفر است. در این حالت A2 و A3 متغیرهای خارج سلولی و تغییرپذیر هستند.
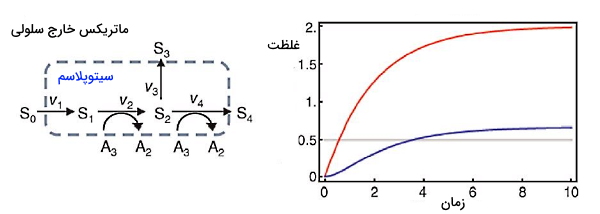
مدلهای تصادفی
در سطح مولکولی واکنشهای بیوشیمایی، واکنشهای تصادفی هستند که تعداد مولکولها را تغییر میدهند. به همین دلیل برای بررسی میکروسکوپی میتوان این واکنشها را فرایندهایی تصادفی در نظر گرفت و تغییر ترکیبات آن را محاسبه کرد. معادلات ریاضی این مدل تغییرات مجزای هر واکنش، تعداد واکنشها در واحد زمان یا تغییرات تصادفی غلظت مواد را توضیف میکند.
مدل بولن
مدل بولن یکی از سادهترین شبکههایی است که پویایی فرایندهای زیستی را مدلسازی میکند. این مدل در شبکهسازی فرایندهایی با حالتهای مجزا در بازه زمانی مستقل به ویژه مسیرهای تنظیم ژن کاربرد دارد. اجزای اسن مدل بر اساس «هست» و «نیست» یا ۰ و ۱ تعرفی میشود. برای مثال ژن فعال (۱) یا غیرفعال (۰) است. این شبکهها از N گره (ژن) تشکیل میشود که K ورودی (برهمکنشهای تنظیمی) و یک خروجی (حالت فعال یا غیرفعال) دارند.
مدل شبکه پتری
شبکههای پتری مدلهای نموداری و محاسباتی هستند که برای شبیهسازی فرایندهای وابسته به زمان استفاده میشوند. بهوسیله این شبکهها میتوان سیستمهای مجزا و موازی را مدلسازی کرد. شبکه پتری از دو جایگاه (گره)، کمانها و انتقالها تشکیل شده میشود. در نمایش نموداری گرهها به شکل دایره نمایش داده میشود و هر گره نمایشدهنده یک نوع مولکول است. مستطیل انتقال نشان نشاندهنده تبدیل شدن یا نشدن و چگونگی تبدیل شدن اجزا به هم است. شکل زیر مدل شبکه پتری برای واکنشهای اولیه گلیکولیز را نشان میدهد. گرههای این مدل مولکولها و انتقالها، واکنشهای آنزیمی مسیر گلیکولیز را نشان میدهد.
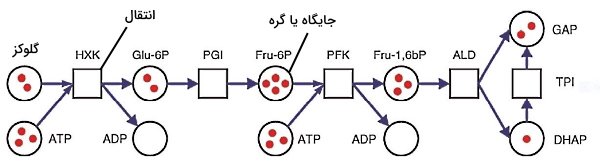
توپولوژی شبکه
بر اساس نوع ارتباط گرهها، یالهای شبکه ممکن است «جهتدار» (Undirected Edges)، «بدون جهت» (Directed Edges) یا «وزندار» (Weighted Edges) باشند. یالهای بدون جهت برای نشان دادن ارتباط ساده بین گرهها استفاده میشود. زمانی از این یالها استفاده میشود که جریان خاصی در شبکه وجود ندارد. این یالها برای رسم شبکههای برهمکنش پروتئین-پروتئین (PPINs) کاربرد دارند.
یالهای جهتدار نوع ارتباط بین گرهها را نشان میدهند. از این یالها برای ترسیم ارتباط سلسله مراتبی استفاده میشود. شبکههای پیامرسانی، تنظیم بیان ژن و واکنشهای متابولیکی را میتوان به کمک این یالها رسم کرد. یک مقدار عددی کنار یالهای وزندار نوشته میشود. این عدد ممکن است فاصله بن دو گره یا پارامترهای دیگری باشد. برای مثال در شبکههای عصبی گرهها نشاندهنده نورون هستند و یالهای وزندار قدرت ارتباط دو نورون را نشان میدهد. این یالها ممکن است جهتدار یا بدون جهت باشند.
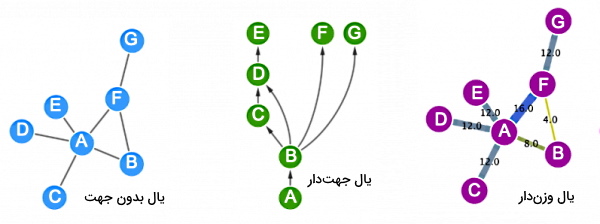
شکل کلی (توپولوژی) شبکه به شیوه اتصال گرهها و یالها بستگی دارد. این توپولوژی به تعداد مولکولهای موجود در سیستم و نوع ارتباط آنها بستگی دارد. درجه، فاصله بین گرهها، اتصال داخل شبکه و مرکزیت گره پارامترهای تعیین توپولوژی شبکه هستند.
- درجه: تعداد یالهای اتصالی به یک گره، درجه شبکه را نشان میدهد. یالهای جهتدار دو عدد درجه (درجه خروجی و درجه ورودی) دارند.
- فاصله بین گرهها: کمترین فاصله بین هر دو گره از شبکه نشاندهنده نوع ارتباط و برهمکنش دو مولکول در شبکه زیستی است.
- اتصال داخل شبکه: اتصال داخل شبکه بین گروهی از گرهها خوشههای شبکه را ایجاد میکند. خوشهها ارتباط نزدیک و برهمکنش زیاد مولکولها را نشان میدهد.
- مرکزیت: مرکزیت جایگاه یک گره بین گرههای دیگر را مشخص میکند و میتوان از ان به عنوان درجه اهمیت گره در شبکه استفاده کرد.
موتیف های شبکه
موتیفها بخشی از شبکههای زیستی هستند که ساختارهای تکراری و شبیه هم دارند. خودتنظیمی مثبت و منفی، آبشارهای مثبت و منفی، لوبهای بازخورد مثبت و منفی، لوبهای پیشخورد، «پروانهای« (Bifan Motif) و «ماژول تکورودی» (Single Input Modules) موتیفهای مدل شبکهای هستند.
بازخورد منفی و مثبت
بازخورد منفی یکی از مکانیسمهای تنظیمی در فرایندهای زیستی است. در این روش یکی از آنزیمهای مسیر سنتز بهوسیله محصول نهایی مهارشده و از تجمع محصول جلوگیری میشود. موتیف فیدبک منفی در شبکههای متابولیکی و رونویسی ژنها وجود دارد. در شکل زیر رابطه بین غلظت و تغییر زمان به شکل شبکهای از گره و خط همراه نمودارهای بیوشیمایی نشان داده شده است. در شکل زیر تولید متابولیت دوم بهوسیله مکانیسم بازخورد منفی غلظت حالت پایدار را کاهش و سرعت ایجاد پاسخ را افزایش میدهد.
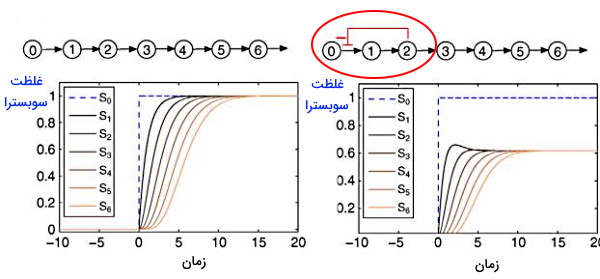
در لوپهای بازخورد مثبت افزایش سنتز مولکول عامل تحریکی ادامه یافتن مسیر سنتز است.
لوپ پیشخورد
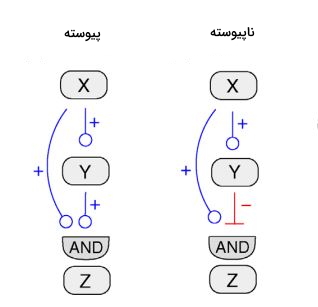
در بسیاری از فرایندهای آناوبولیکی بدن سنتز یک ترکیب، سنتز ترکیب بعدی را کنترل میکند. به این مکانیسم تنظیمی لوپ پیشخورد گفته میشود. شکل زیر موتیفی از شبکه شامل سه ژن Z، X و Y را نشان میدهد. در این موتیف پروتئین ژن X، ژن Z را مستقیم و ژن Y را غیرمستقیم تنظیم میکند.
ماژول تکورودی
در ماژول تکورودی یک مولکول در سیستم فعالیت تعداد زیادی زا مولکولها را کنترل میکند. بیان آنزیمهای متابولیسمی بهوسیله این مدل کنترل میشود. در این روش ژن بهوسیله یک فاکتور رونویسی اصلی فعال میشود و پروتئین آن چند آنزیم مجزا را فعال میکند.
موتیف پروانه ای
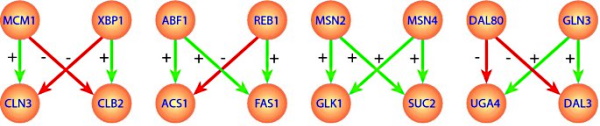
موتیف پروانهای بیشتر در شبکههای تنظیم بیان ژن و پیامرسانی سلولی وجود دارد. در این موتیف از دو گره اصلی و دو گره تنظیم شونده تشکیل شده است. هر گره اصلی با دو گره تنظیم شونده ارتباط دارد. یالهای این دو گره با هم تقاطع دارند و شکلی شبیه بال پروانه ایجاد میکنند. برای مثال دو پروتئین کینازی که در مسیر تنظیم بیان ژن دو فاکتور رونویسی را فسفوریله و فعال یا مهار میکنند، در شبکه با موتیف پروانهای مدلسازی میشوند.
مدل دستوری
در مدل دستوری وضعیت هر یک پارامترهای سیستم بر اساس مجموعهای از قوانین و دستورات به روز میشود. برای مثال میتوان دستور مدل را اینگونه تعریف کرد که پروتئین Y در صورتی فسفوریله میشود که کیناز X فعال شود و مهارکننده Z در سلول وجود نداشته باشد. در این مدل تمام تغییرات احتمالی حالتها در نظر گرفته میشود، اما تمام حالتهایی ممکن است اتفاق بیفتد، در نظر گرفته نشده است.
مدل آماری زیست شناسی سامانه ای
مدلهای آماری بین متغیرهای اندازهگیری شده در آزمایشهای زیستشناسی ارتباط برقرار کرده و مسیری برای استخراج ارتباط بین فرایندها به وسیله فرمولهای ریاضی ایجاد میکند. بهوسیله این مدلها میتوان مقادیر عددی پارامترها را دستهبندی و ارتباط بین آنها را مشخص کرد. ANOVA، «توزیع گوسین» (Gaussian distribution) و «توزیع پواسون» (Poisson distribution) ازجمله محاسبات آماری هستند که در مدلسازیهای زیست شناسی سامانه ای از آنها کمک گرفته میشود.
مسیر های زیستی مدلسازی
متابولیسم، انتقال پیام، فرایندهای زیستی ویژه و بیان ژن فرایندهای اصلی سیستمهای زنده هستند که بهوسیله مدلسازیهای زیست شناسی سامانه ای میتوان رابطه بین اجزای آن را بررسی و نتیجه تغییر ساختار یا عملکرد هر بخش در کل سیستم را بررسی کرد.
- متابولیسم: موجودات زنده به انرژی و ماده برای انجام فرایندهای زیستی، ادامه حیات و سنتز سلول جدید نیاز دارند. فرایند تامین انرژی و سنتز ساختارهای جدید متابولیسم نام دارد. متابولیسم از دو دسته واکنشهای آنزیمی کاتابولیکی (تجزیه) و آنابولیکی (سنتز) تشکیل شده است. بنابراین در متابولیسم ترکیبات A به وسیله آنزیمها به ترکیبات B تبدیل میشوند. در نتیجه غلظت ترکیبات و سرعت پارامترهای مدلسازیهای متابولیکی هستند.
- انتقال پیام: در سیستمهای زنده سلولها انتقال پیامهای مولکولی روش اصلی برقراری ارتباط بین سلولها است. پیام این مولکولها فعالیت سلولها و فعالیت سلولها فعالیتهای سیستم یکپارچه موجود زنده را تغییر میدهد. هورمونها، فورمونها، گرما، سرما، تغییر فشار اسمزی، نور، تغییر جریان الکتریکی و تغییر غلظت متابولیتها پیامهای تغییردهنده فعالیت سلول است. پارامترهای مدلسازی این فرایند سنتز و تجزیه ترکیبات، تغییرات مولکولی (فسفوریلاسیون، اسیلاسیون و متیلاسیون) و مهار یا فعال شدن واکنشهای بیوشیمیایی هستند. چهار تفاوت بین انتقال پیام و متابولیسم در مدلسازی این دو فرایند تفاوت ایجاد میکند.
- در متابولیسم جرم اما در پیام رسانی اطلاعات منتقل میشود.
- مولکولهای اصلی در متابولیسم تعداد زیادی آنزیم و سوبسترا اما در انتقال پیام انواع مولکولهای سلولی (گیرنده، نوکلئوتید، آنزیم، فاکتورهای رونویسی و ژنها) شرکت میکنند.
- تعداد مولکولها در متابولیسم بسیار بیشتر از تعداد مولکولهای پیامرسانی است.
- غلظت آنزیمها در متابولیسم بسیار کمتر از سوبسترا است و واکنش بیوشیمیایی به حالت نیمهپایدار میکائیلیس منتن میرسد. اما نسبت غلظت آنزیم و سوبسترا در پیامرسانی تقریبا یکسان است.
- فرایندهای زیستی ویژه: فرایندهای زیستی که در مدلسازیهای زیست شناسی سامانه ای از آنها استفاده میشود مجموعهای از واکنشهای پیچیده یا واکنشهایی هستند که با دیگر واکنشها برهمکنش دارند. چرخه سلولی و افزایش سن دو فرایند مدلسازی هستند.
- بیان ژن: تمام فرایندهای زیستی مولکولهای ساختاری در سیستمهای پروکاریوتی و یوکاریوتی بهوسیله ژنها کنترل میشود. به همین دلیل مدلسازی و پیشبینی روند تغییرات آنها نقش مهمی در شناخت سامانههای زیستی و درمان بیماریها دارد. از دو روش برای مدلسازی بیان ژن استفاده میشود.
- برهمکنشهای مولکولی (DNA-فاکتور رونویسی، RNA-آنزیم پلیمراز و مهارکننده یا فعالکننده-RNA) و تنظیمهای بازخوردی یک یا تعداد کمی از ژنها مدلسازی شود.
- پروفایل (سطح بیان در زمانهای مختلف) و الگوی (تغییر بیان ژن در شرایط آزمایشگاهی مختلف) بیان همزمان چند ژن بهوسیله آرایههای DNA بررسی شود.
نرم افزار های زیست شناسی سامانه ای
بر اساس نوع مدلهای زیست شناسی سامانه ای میتوان از نرمافرازهای متفاوتی برای انجام محاسبات ریاضی و رسم نمودارهای گرافیکی بهره برد. با استفاده از این نرمافزارها زمان لازم برای انجام محاسبات کاهش یافته اما دقت اطلاعات دریافتی افزایش مییابد. «متلب» (MATLAB)، «متمتیکا» (Mathematica)، «آر» (R)، «ورچوآل سل» (Virtual Cell) و «گرومکس» (GROMACS) نرمافزار زیست شناسی سامانه ای هستند که د راین بخش از مجله فرادرس کاربرد آن ها را توضیح میدهیم.
نرم افزار MATLAB
متلب نرمافزاری بر پایه کدنویسی و مدلسازی دستوری است برای انجام محاسبات، آنالیز دیتای و شبیهسازی حوزههای مختلف است. از الگوریتمهای این نرمافزار میتوان برای شبیهسازی سیستمهای زیستی، آنالیز تصاویر DICOM سیستمهای تصویربرداری پزشکی، آنالیز تصاویر میکروسکوپی، انجام محاسبات سینتیکی و رسم نمودارهای زیستی کمک گرفت.
نرم افزار Mathematica
Mathematica نرم افزاری برای انجام محاسبات پیچیده ریاضی و رسم نمودار است. معادلات این نرمافزار برای تحلیل دیتای پژوهشهای مهندسی، جغرافیا، آماری، اقتصادی و زیستشناسی کاربرد دارد.
Mathematica به شکل دو نسخه آنلاین و دسکتاپ در اختیار کاربران قرار داده شده است. این نرمافزار امکان آنالیز تصاویر DICOM سیستمهای تصویربرداری پزشکی، تصاویر میکروسکوپهای نوری و الکترونی، نمودارهای ثبت جریان الکتریکی بدن (الکتروکاردیوگرام، الکترومیوگرام یا الکتروآنسفالوگرام) را آنالیز یا محاسبات مدلسازیهای بیوشیمیایی را انجام داد.
نرم افزار R
R نرمافزاری رایگان برای انجام محاسبات آماری و رسم نمودارهای گرافیکی است. این نرمافزار آفلاین به شکل نسخههای ویندوز، لینوکس و MacOS برای کاربران طراحی شده است. این نرمافزار امکان وارد کردن معادلات جدید به سیستم را با استفاده از زبانهای برنامهنویسی برای کابر فراهم میکند.
نرم افزار Virtual Cell
Virtual Cell نرمافزاری کاربردی و رایگان برای مدلسازیهای زیستشناسی مولکولی است که نسخههای ویندوز، MacOS و لینوکس آن را میتوان از وبسایت رسمی نرمافزار دانلود کرد. این نرمافزار امکان آنالیز و مدلسازی نتایج آزمایشگاهی و فرضیات اثبات نشده را فراهم میکند. تبدیل خودکار اطلاعات و روابط زیستشناسی به معادلات ریاضی یکی از ویژگیهایی است که این نرمافزار را به گزینهای مناسب برای افراد مبتدی تبدیل میکند.
دیتای نرمافزار Virtual Cell در سه گروه «مدل زیستی» (BioMolel)، «مدل ریاضی» (MathModel) و «شکل فضایی» (Geometry) طبقهبندی شده است.
- BioMolel: مدل زیستی بخش گرافیکی نرمافزار است که از توضیح فیزیولوژی مدل (ساختار زیستی، مولکولها، واکنشهای بیوشیمیایی و مسیر متابولیکی) و شرایطی است که معادله ریاضی برای آن تعریف میشود.
- MathModel: مدل ریاضی بخش محاسباتی نرمافزار است که معادلات لازم را با استفاده از مدلزیستی یا دستورات وارد شده بر اساس زبان VCell Marup در اختیار کاربر قرار میدهد.
- Geometry: این بخش امکان انتخاب طراحی مدلهای تکبعدی، دوبعدی یا سهبعدی را برای کاربر فراهم میکند.
نرم افزار GROMACS
گرومکس یکی از نرمافزارهای شبیهسازی دینامیک مولکولی در زیستشناسی سامانه ای است. این نرمافزار معادلات حرکت نیوتونی را برای مولکولها و برهمکنش آنها با هم شبیهسازی میکند. این نرمافزار رایگان بر اساس مدلسازیهای دستوری و برای سیستم عامل لینوکس طراحی شده است. در زیست شناسی سامانه ای از این نرمافزار برای آنالیز برهمکنش سیستمهای زیستی، شبیهسازی غشای دولایه، شبیهسازی آنزیمها و طراحی سیستمهای دارویی بهینه استفاده میشود.
جمع بندی
در این مطلب از مجله فرادرس توضیح دادیم که زیست شناسی سامانهای موجود زنده را یک سیستم یکپارچه در نظر میگیرد و از روابط ریاضی، نتایج آزمایشهای تجربی و نرمافزارهای کامپیوتری برای توضیح و پیشبینی رفتار این سیستم استفاده میکند. مدلهای این روش بر پایه مسیرهای تنظیم بیان ژن، متابولیسم و انتقال پیام به شکل شبکهای، آماری و دستوری با استفاده از نرمافزارهای مختلف طراحی میشوند.