آموزش نرم افزار ژن رانر – به زبان ساده + نحوه آنالیز نتایج

بررسی ژنوم و پروتئینهای موجودات زنده به شناخت ویژگیهای منحصر به فرد و رابطه تکاملی بین این موجودات کمک فراوانی کرده است. به علاوه با شناخت ساختار و ویژگیهای این مولکولهای حیاتی میتوان روشهای تشخیصی و درمانی جدید برای بیماریهای انسانی، جانوری و گیاهی طراحی کرد. اضافه شدن نرمافزارها و الگوریتمهای محاسباتی به زیستشناسی زمان و انرژی لازم برای این تحقیقات را بسیار کاهش داده است. نرم افزار ژن رانر یکی از ابزارهایی است که به کمک آن میتوان ویژگیهای اسید نوکلئیکها و پروتئینها را بررسی کرد. در این مطلب از مجله فرادرس قابلیتهای این نرمافزار در طراحی پرایمر و کلونینگ را توضیح میدهیم.
نرم افزار ژن رانر چیست ؟
«ژن رانر» (Gene Runner) یکی از نرمافزارهای آفلاین آنالیز توالیهای نوکلئوتیدی و آمینواسیدی است. این نرمافزار امکان بررسی جایگاه برش «آنزیمهای محدودکننده» (Restriction Enzyme) در ژن و طراحی پرایمر برای تست PCR فراهم میکند. این نرمافزار به شکل رایگان در اختیار تمام کاربران قرار دارد و نسخه ویندوز (۳۲ و ۶۴ bit) را میتوان از وبسایت رسمی نرمافزار دانلود کرد. برای نصب این نرمافزار کافی است فایل exe آن را در سیستم اجرا کنید.
رابط کاربری نرم افزار ژن رانر
رابط کاربری ژن رانر کاملا گرافیکی است و برای استفاده از آن به کدنویسی نیاز ندارید. با کلیک بر آیکون برنامه صفحهای شبیه تصویر زیر برای شما باز میشود. بالای این صفحه یک نوار ابزار اصلی و یک نوار کلیدهای میانبر قرار دارد.
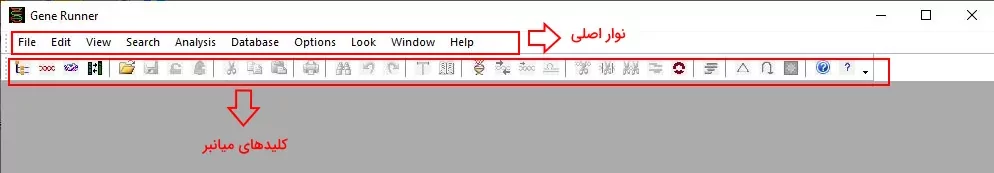
گزینههای نوار کلیدهای میانبر را میتوانید با استفاده از فلش انتهای راست تغییر دهید. «فایل» (File)، «ویرایش» (Edit)، «جست و جو» (Search)، «نمایش» (View)، «آنالیز» (Analysis)، «پایگاهداده» (Database)، «گزینهها» (Options)، «نما» (Look)، «پنجره» (Window) و «راهنما» (Help) آیتمهای نوار ابزار اصلی هستند.
- فایل: در این بخش میتوانید فایل توالیهای نوکلئوتیدی یا آمینواسیدی که از پایگاه دادههای مختلف با فرمت SQE دانلود کردهاید را با استفاده از کلید Open در نرمافزار باز کنید. یا با استفاده از کلید New صفحه جدیدی برای وارد کردن توالی نوکلئوتیدی، آمینواسیدی یا همترازی (DNA، RNA یا پروتئین) باز کنید. پس از اتمام پروژه با استفاده از Save میتوانید تغییرات ایجاد شده را ذخیره نمایید.
- ویرایش: با استفاده از گزینههای نرمافزار میتوانید بخشی از توالی انتخاب شده را حذف، در صفحه دیگری کپی یا نامگذاری کنید. با استفاده از گزینه "Geometry" در بخش میتوانید تعداد رشتههای نمایشی (اسید نوکلئوئیک تکرشتهای یا دورشتهای) و توپولوژی توالی (اسید نوکلئوئیک خطی یا حلقوی) را تغییر دهید.
- نمایش: با استفاده از گزینه Translation (ترجمه) در این بخش نرمافزار توالی آمینواسیدی متناظر با کدونها را زیر توالی نوکلئوتیدی نشان میدهد. گزینه "Map" (نقشه) جایگاه کل توالی ژنتیکی در کروموزوم را نشان میدهد.
- جست و جو: در این بخش از نوار ابزار میتوانید بهراحتی موتیفهای نوکلئوئیکاسید و پروتئینی مورد نظر خود در توالی جست و جو کنید.
- آنالیز: در این بخش میتوانید جایگاه آنزیمهای مهارکننده در توالی نوکلئوتیدی، انواع پروپ پروتئینی و همترازی DNA-DNA یا پروتئین-پروتئین را بررسی کنید. آنالیز Oligo ویژگیهای توالی انتخابی ازجمله محتوای GC و دمای ذوب را نشان میدهد.
- پایگاهداده: در این بخش به جدول پیشفرض آمینواسیدها، آنزیمهای محدودکننده، پپتیدازها، موتیفهای پروتئینی و پروتئینهای مهارکننده وجود دارد. به علاوه کاربر میتواند جدول دلخواه خود را طراحی کند.
- گزینهها: با کلیک بر گزینه "Style" در این بخش میتوانید رنگ و فونت توالیها را تغییر دهید. گزینه "Default Directories" در این بخش محل ذخیره فایلها در سیستم را نشان میدهد.
- نما: با استفاده از این گزینه میتوانید ویژگیهای ظاهری نوار ابزارها را تغییر دهید.
- پنجره: به کمک این گزینه میتوانید در نرمافزار پنجره جدید باز کنید یا اندازه پنجرهها را تغییر دهید.
- راهنما: در این بخش اطلاعات مربوط به ویژگیهای نرمافزار و شیوه کار بخشهای مختلف نوشته شده است.
آنالیز اسید نوکلئوئیک در ژن رانر
از نرمافزار ژن رانر میتوان برای آنالیز آنزیمهای محدودکننده، ایجاد جهش جهتدار، توالییابی پرایمرها و پروپهای هیبریداسیون، طراحی پرایمرهای PCR، تعیین دما ذوب و الگوی خواندن باز (Open Reading Frame | ORF) کمک گرفت.
آنالیز آنزیم محدودکننده
در مطالعات کلونینگ و هیبریداسیونهای اسید نوکلئیک از آنزیمهای محدودکننده برای برش ژن اصلی استفاده میشود. به همین دلیل برای پیشگیری از برش نوکلئوتیدهای داخلی پرایمر بهوسیله این آنزیمها بررسی جایگاه برش این آنزیمها در توالی طراحی شده ضروری است. بهوسیله گزینه Cut in و Cut out در آنالیزهای ژن رانر میتوان جایگاه برش آنزیمهای محدودکنندهای که در جدول دیتابیس آنزیمهای محدودکننده هست را در توالی ژنی مورد نظر بررسی کرد. این آنالیز را میتوان با در نظر گرفتن پارامترهای غلظت یونهای محیط، حساسیت به آنزیم متیلاز، طول توالی شناسایی و انتهای چسبنده برشها انجام داد.
این آنالیز را میتوانید با جدول دلخواه خود انجام دهید. برای این منظور در پنجره باز شده آنالیز روی گزینه "Find Table" (پیدا کردن جدول) کلیک کرده و فایل جدول را انتخاب کنید. گزینه "cut sites#" در این پنجره حداقل و حداکثر تعداد برش در توالی ژنی را نشان میدهد. پیشفرض این گزینه بین ۱ یا ۱۰ است. در نتیجه، در آنالیز آنزیمهایی بررسی میشوند که حداقل یک برش و حداکثر ۱۰ برش در ژن ایجاد میکنند. برای پیدا کردن آنزیمهایی که جایگاه برش در توالی مورد نظر ندارند باید خروجی را به "List non-cutters" تغییر دهید.
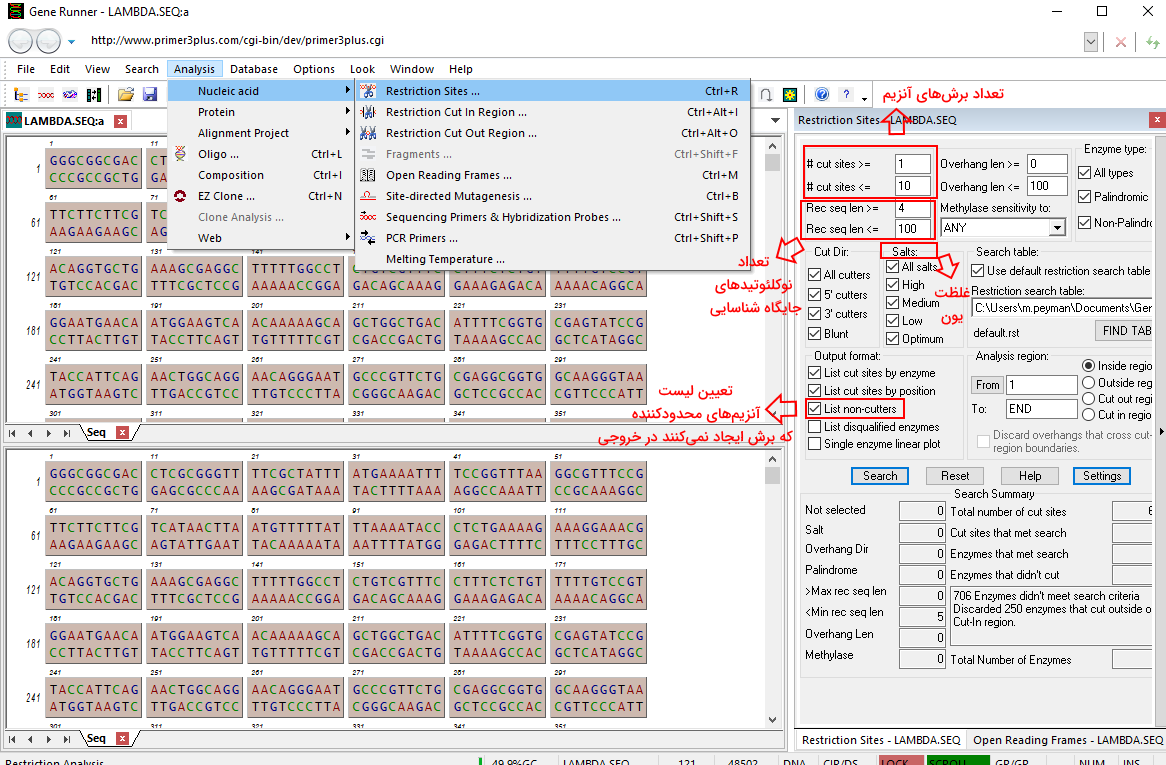
گزینه "Rec seq len" طول توالی شناسایی آنزیمها را نشان میدهد. آنزیمهایی که طول تولی شناسایی بیشتری دارند، برشهای کمتری در ژن ایجاد میکنند. مقدار پیشفرض این گزینه بین ۰ و ۱۰۰ تنظیم شده است. گزینه "Overhang len" در این پنجره طول چسبنده دو توالی ایجاد شده پس از برش آنزیم محدودکننده را نشان میدهد. اگر مقدار حداقل این گزینه را بیشتر از 0 قرار دهیم، آنزیمهایی که پس از برش انتهای صاف ایجاد میکنند از آنالیز حذف میشوند. "Methylase sensitivity" (حساسیت به متیلاز)، "Enzyme Type" (نوع آنزیم)، "Cut Direction" (جهت برش | Cut Dir)، "Output format" (فرمت خروجی)، "Restriction map" (نقشه برش) و "Analysis region" (ناحیه آنالیز) گزینههای دیگری هستند که به کمک آنها پارامترهای مختلف آنالیز را تعیین می کنیم.
- حساسیت به متیلاز: به کمک این گزینه میتوان آنزیمهای محدودکننده حساس به آنزیم متیلاز را حذف (ANY) یا نوع آنزیم متیلاز را تعیین کرد. انتخاب گزینه "none" فیلتر حساسیت به متیلاز را در آنالیز اعمال نمیکند.
- نوع آنزیم: آنزیمهای محدودکننده بر اساس نوع توالی شناسایی به دو نوع پالیندرومی (Palindromic) و پالیندرومی (Non-Palindromic) تقسیم میشوند. در توالیهای پالیندرومی اگر دو رشته همجهت خوانده شود، توالی نوکلئوتیدها یکسان است. برای مثال توالی یک رشته $$5^\prime TTAGCACGTGCTAA 3^\prime$$ و رشته دیگر $$3^\prime AATCGTGCACGATT 5^\prime$$ است. در این بخش میتوان اثر یک گروه از این آنزیمها یا هر دو (All Types) را بر توالی ژنی بررسی کرد.
- جهت برش: در این بخش آنزیمها را بر اساس انتهای برش ایجاد شده (انتهای $$5^\prime$$ چسبنده ، انتهای $$3^\prime$$ چسبنده ، انتهای صاف یا همه) انتخاب میکنیم.
- فرمت خروجی: در این بخش تعیین میکنیم چه اطلاعاتی (جایگاه برش، نوع آنزیم، نوع انتها، آنزیمهای نامناسب و نمودار برش هر آنزیم) فایل خروجی آنالیز گزارش شود.
- ناحیه آنالیز: در این بخش نقطه شروع و پایان توالی نوکلئوتیدی تعیین میشود. گزینه "Within region" آنزیمهایی را نشان میدهد که جایگاه شناسایی و برش آنها در ناحیه انتخاب شده است. گزینه "Outside region" آنزیمهایی را نشان میدهد که توالی شناسایی آنها کاملا در ناحیه انتخاب شده نیست. "Cut out region" آنزیمهایی را نشان میدهد که جایگاه برش آنها خارج از ناحیه انتخاب شده است. بخشی از توالی شناسایی این آنزیمها در ناحیه انتخاب شده و بخشی از آن خارج از این ناحیه قرار دراد. "Cut in region" جایگاه برش این آنزیمها کاملا در ناحیه انتخاب شده قرار دارد و خارج از این محدوده ژن را برش نمیدهند. نتیجه این آنالیز به طراحی وکتور کمک میکند.
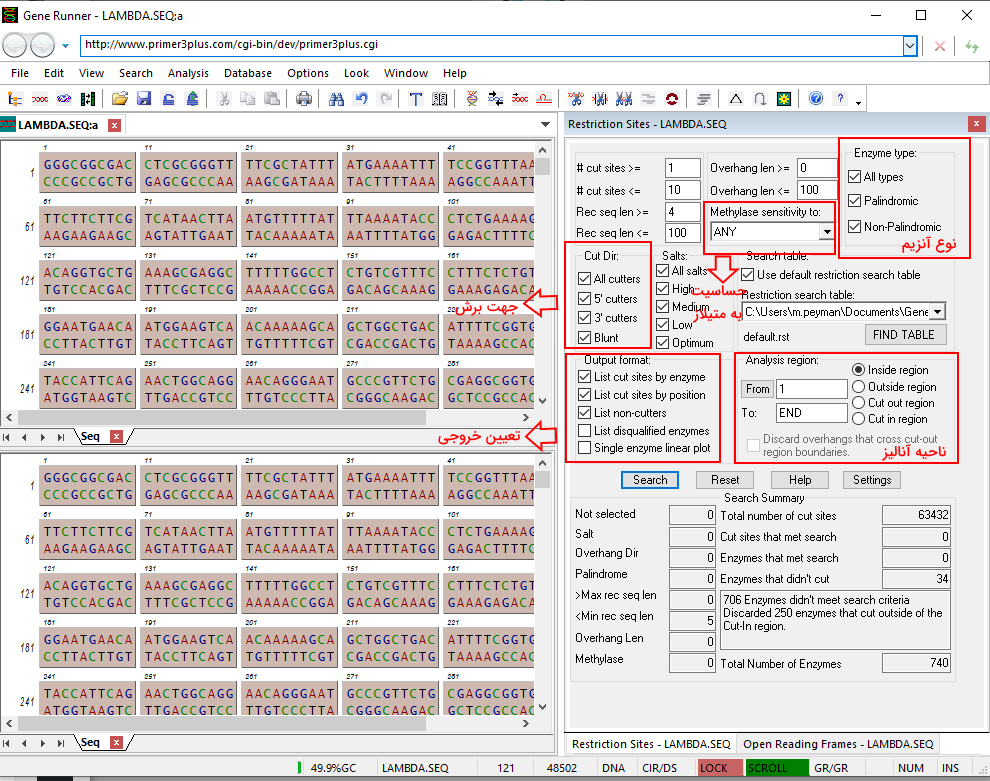
پس از انجام آنالیز نرمافزار سه گزارش بر اساس نوع آنزیم محدودکننده، جایگاه برش آنزیم و آنزیمهایی که برشی ایجاد نکردهاند را در اختیار کاربر قرار میدهد. همچنین در بخش پایینی تعداد کل آنزیمهای بررسی شده، تعداد کل جایگاههای برش، تعداد جایگاههای بررسی که با پارامترهای تعیین شده همخوانی داشتهاند، تعداد آنزیمهایی که در توالی هدف برش ایجاد میکنند، تعداد آنزیمهایی که برش ایجاد نمیکنند و تعداد آنزیمهایی که با پارامترهای تعیین شده همخوانی ندارند، گزارش میشود.
آنالیز الگوی ژن
خواندن الگوی باز (ORFs) ناحیه از ژن بین کدون شروع و کدون پایان است. برای پیدا کردن OFRs در نرم افزار ژن رانر، توالی ژنی مورد نظر باید در پنجره فعال باز باشد. برای باز کردن پنل این آنالیز میتوانید از کلید میانبر Ctrl + M یا مسیر Analysis > Nucleic Acid > Open Reading Frames استفاده کنید. "ORF"، "ORF size"، "Frame control"، "translation Control"، "ORF Control" و "Analysis region" بخشهای این پنل هستند که بهوسیله آن پارامترهای آنالیز ORF را تغییر میدهیم.
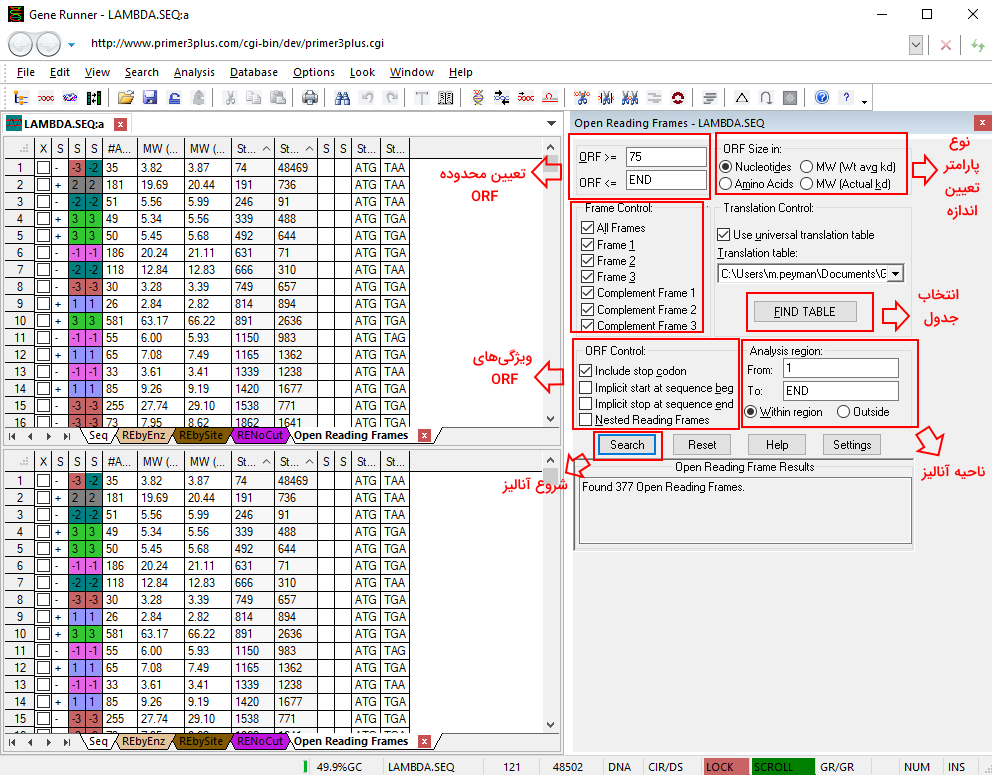
- ORF: در این بخش حداقل و حداکثر اندازه ORF تعیین میشود. حداقل تعداد پیشفرض OFR ۷۵ باز در نظر گرفته شده است. جست و جوی ORF در ژنوم حلقوی با رسیدن به کدون پایان مشخص توقف مییابد اما در ژنوم خطی تا انتها ادامه دارد. با تغییرات تعداد حداقل و حداکثر این گزینه میتوان تعداد ORF پروتئین را بررسی کرد.
- ORF size in: این بخش نوع پارامتر تعیینکننده اندازه ORF را مشخص میکنیم. اندازه ORF بر اساس تعداد نوکلئوتیدها، آمینواسیدها یا میانگین وزن مولکولی تعیین میشود. میانگین وزن مولکولی در آنالیزهای همراه ترجمه پروتئین کاربرد دارد.
- Frame Control: در این بخش نقطه شروع ترجمه و آنالیز ORF تعیین میشود. با انتخاب گزینه "Frame 1" ترجمه از اولین، با انتخاب گزینه "Frame 2" ترجمه از دومین و با انتخاب گزینه "Frame 3" ترجمه از سومین باز توالی ژنی شروع میشود. با انتخاب Complementary frame 1 ترجمه از آخرین باز مکمل، با انتخاب "Complementary frame 2" ترجمه از دومین باز مکمل از انتها و با انتخاب Complementary frame 3 ترجمه از سومین باز مکمل انتهای توالی ژنی شروع میشود. با انتخاب All Frames آنالیز برای تمام گزینهها انجام میشود.
- ORF Control: در این بخش ویژگیهای ORF بر اساس ترجمه کدون پایان (Include stop codon)، شروع ترجمه از کدون آغاز (implicit start at sequence beginning)، توقف ترجمه در کدون پایان ( implicit stop at sequence end) و الگوی خواندن آشیانهای (Nested Reading Frames) تعیین میشود. با انتخاب الگوی خواندن لانه تمام ORFs احتمالی با کدون پایان یکسان و کدون آغاز متفاوت، در آنالیز پایانی گزارش میشود.
- Translation Control: در این بخش جدول آمینواسید برای ترجمه توالی نوکلئوتیدی انتخاب میشود و کاربر میتواند از بخش Find Table جدول مورد نظر خود را انتخاب کند.
- Analysis region: در این بخش محدوده آنالیز توالی ژنی مشخص میشود.
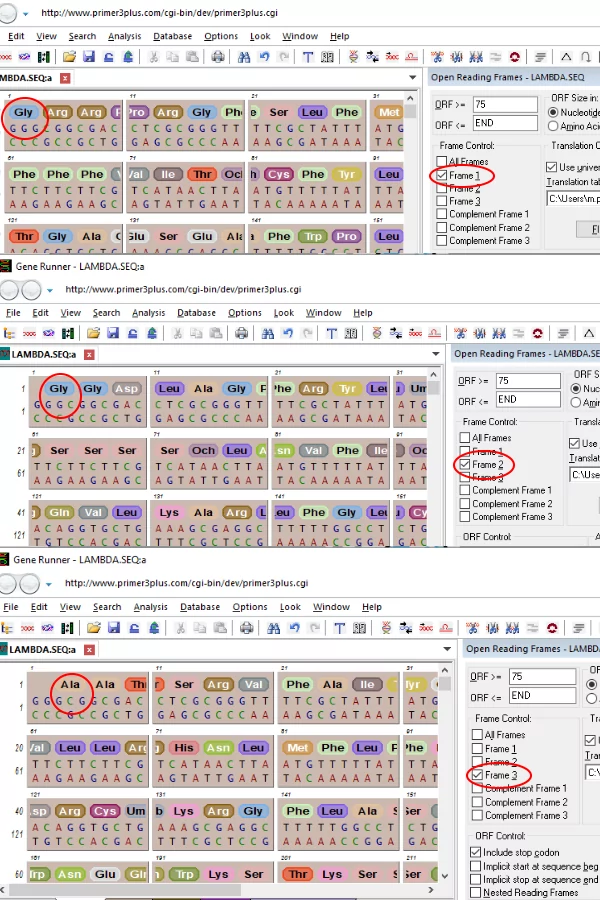
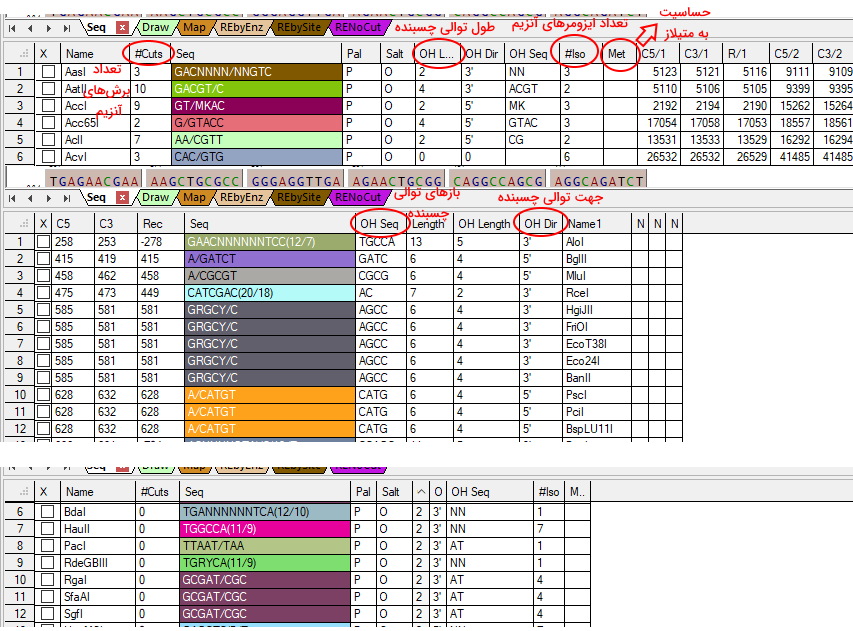
پس از انجام آنالیز در بخش پایینی پنل تعداد الگوهای خواندن باز در توالی انتخاب شده و در پنجره سمت چپ نوع رشته DNA (مثبت و منفی)، الگوی شروع و پایان، شماره کدون شروع و پایان، نوع کدون شروع و پایان، میانگین وزن مولکولی آمینواسیدها و تعداد آمینواسیدهای الگو گزارش میشود.
طراحی پرایمر جهش زایی در ژن رانر
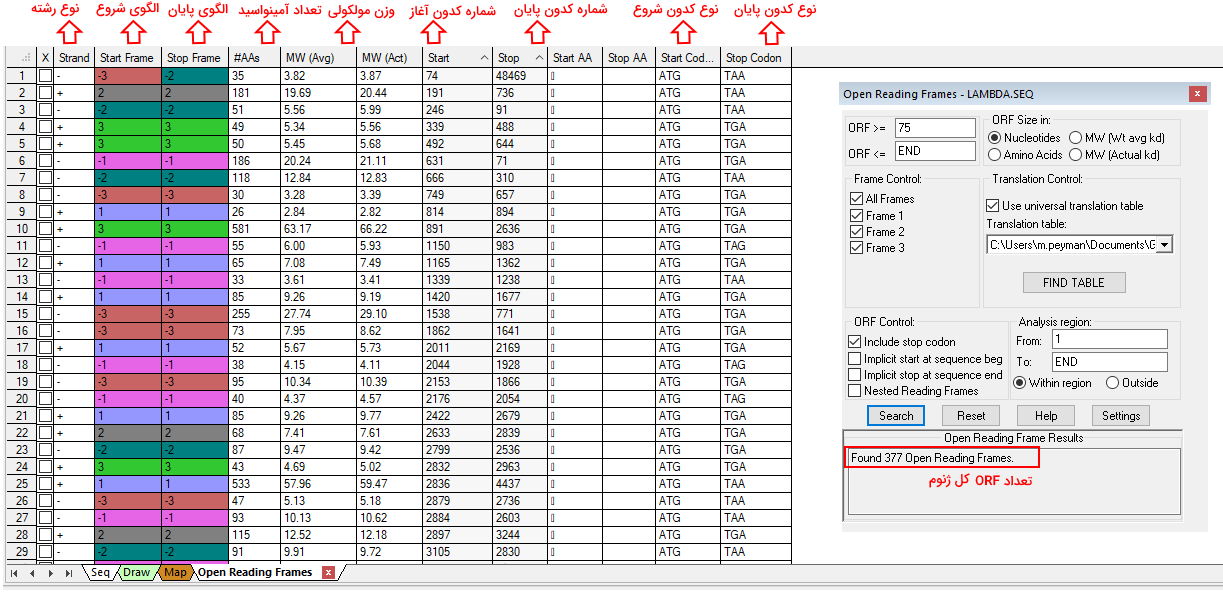
برای شروع آنالیز جهشزایی از کلید میانبر Ctrl + B یا مسیر Analysis > Nucleic Acid > Site-directed Mutagenesis استفاده کنید. بهوسیله این آنالیز میتوان پرایمرهای اولیگونوکلئوتیدی برای ایجاد جهشهای نوکلئوتیدی جهتدار طراحی کرد. تعداد نوکلئوتیدهای تغییریافته، درصد نوکلئوتیدهای C و G، دمای ذوب و تشکیل نشدن ساختار دوم ویژگیهای پرایمر مناسب برای ایجاد جهشهای آمینواسیدی یا جایگاه اتصال و برش آنزیم محدودکننده است. Old Nts، Old Aas و New Aas سه بخش اصلی پنل آنالیز جهشزایی نرم افزار ژن رانر است که توالی نوکلئوتیدی را مشخص میکند.
- Old Nts: در این بخش توالی نوکلئوتیدی اصلی (با هایلات نوکلئوتیدهای ژنوم هدف یا تایپ توالی) نوشته میشود. توالی نوکلئوتیدی این قسمت باید شامل بازهای دو رشته باشد. اگر DNA تکرشتهای یا RNA را در اختیار دارید با استفاده از گزینه Geometry بخش ویرایش نوار ابزار، DNA دو رشتهای ایجاد کنید. این حداکثر ۱۰۰ نوکلئوتیدی است.
- Old Aas: در این بخش توالی آمینواسیدی (حداکثر ۳۳ آمینواسید) ژن انتخابی با نماد تکحرفی نشان داده میشود. با استفاده از نوار کشویی Frame در این بخش، نوکلئوتید شروع ترجمه تغییر میکند. آمینواسیدهای این بخش را نمیتوان تغییر داد.
- New Aas: در این توالی آمینواسیدی مورد نظر پس از ایجاد جهش وارد میشود. به جای آمینواسید حذف شده از - استفاده کنید. برای مثال برای حذف تروئونین از توالی ATYDS، توالی A-YDS را در کادر New Aas وارد کنید. برای جایگزین کردن آمینواسید نماد تکحرفی آمینواسید جدید را با قبلی عوض کنید استفاده کنید. برای مثال برای تغییر دومین گلایسن به تروئونین در توالی G G D L A G به تروئونین، G Y D L A G را وارد کادر کنید.
هدف جهش بر اساس تغییرات ایجاد شده در کادر New Aas یا بخش آنزیم محدودکننده به صورت خودکار بهوسیله نرمافزار تعیین میشود. با انتخاب گزینه پیدا کردن حذف و اضافه جایگاه آنزیم محدودکننده (Find RE site additions & deletions) تمام نوکلئوتیدهای طراحی شده با جدول آنزیمهای محدودکننده چک و با توالی اصلی مقایسه میشود. با انتخاب گزینه اضافه کردن جایگاه آنزیم محدودکننده (Add RE site) و انتخاب نوع آنزیم از نوار کشویی، یک جایگاه برش آنزیم انتخاب شده به توالی اصلی اضافه میشود. برای پیشگیری از تغییر آمینواسیدها پس از اضافه شدن جایگاه آنزیم محدودکننده حتما کلید "Preserve translation" را فعال نگه دارید.
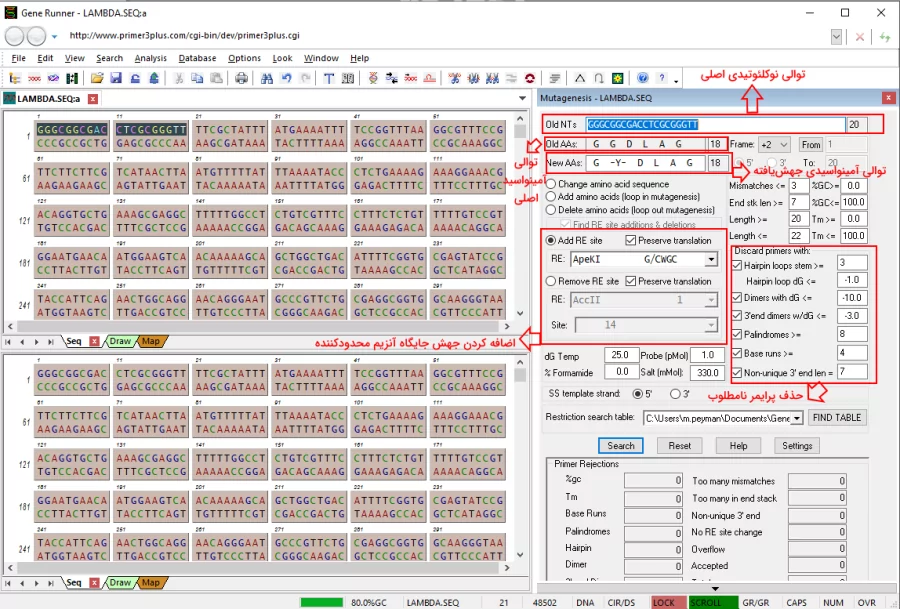
در بخش بالا و سمت راست پنل آنالیز جهشزایی میتوانید خصوصیات پرایمر مطلوب را انتخاب کنید.
- گزینه "Mismatches" تعداد حداکثر بازهای غیرمکمل در پرایمر را نشان میدهد. تعداد کم بازهای غیرمکمل احتمال ایجاد جهش مورد نظر را افزایش میدهد. افزایش این تعداد اختلاف دمای ذوب ($$T_m$$) پرایمر و توالی هدف را افزایش میدهد. این گزینه در پیشفرض نرمافزار روی ۳ جفت غیرمکمل تنظیم شده است و به ازای اضافه شده هر آمینواسید به شکل خودکار ۳ جفتباز به آن اضافه میشود.
- در کادر End stk len تعداد بازها در دو انتهای جهش را تعیین کنید. تعداد این بازها با افزایش طول جهش افزایش مییابد. این بازها برای افزایش پایداری هیبریداسیون پرایمر با ژن هدف ضروری است. عدد این پارامتر در پیشفرض نرم افزار ژن رانر ۷ باز در نظر گرفته شده است. توالی کمتر از ۴ باز، کارایی هیبریداسیون پرایمر و توالی هدف را کاهش میدهد.
- با استفاده از دو گزینه "Length" حداکثر و حداقل طول پرایمر را تعیین کنید. پرایمرهای کوتاهتر با دقت بیشتری به پرایمر هدف متصل میشوند. سنتز پرایمرهای بلندتر هزینه پیشتری به پروژه تحمیل میکند و دقت اتصال را کاهش میدهد. مقدار پیشفرض حداقل طول پرایمر بهوسیله نرم افزار ژن رانر برابر با تعداد نوکلئوتیدهای توالی اصلی در نظر گرفته میشود. این مقدار را افزایش ندهید.
- با استفاده از Tmحداکثر و حداقل دمای ذوب پرایمر را تعیین کنید. اگر دمای ذوب بسیار پایین کارایی اتصال پرایمر را کاهش و دمای ذوب بسیار بالا احتمال اتصال پرایمر به توالی اشتباه را افزایش میدهد.
- در کادرهای GC درصد حداکثر و حداقل بازهای G و C در پرایمر را تعیین کنید. افزایش این درصد با افزایش دمای ذوب، اتصال پرایمر به توالیهای اشتباه و تشکیل ساختارهای سنجاقسری همراه است.
بخش "Discard Primer With" امکان تعیین خصوصیات نامطلبوب پرایمر را فراهم میکند. پرایمرهایی دارای این ویژگیها (تشکیل لوپهای سنجاقسری، تشکیل لوپهای G، تشکیل دیمرهای نوکلئوتید G، دیمرهای انتهای $$3^\prime$$، توالی مکمل، انتهای $$3^\prime$$ غیر منحصر به فرد) از گزارش پایانی آنالیز حذف میشود.
- تشکیل لوپهای سنجاقسری در پرایمر احتمال ایجاد جهش را کاهش میدهد. به همین دلیل کمترین عدد برای تشکیل پایه لوپ به شکل پیشفرض در این بخش درنظر گرفته شده است.
- افزایش پایداری لوپ G احتمال ایجاد جهش را افزایش میدهد. مقادیر زیر 0 این پارامتر سبب تشکیل اولیگونوکلئوتیدهای بهینه میشود.
- حداکثر آستانه پایداری را برای پیشگیری از تشکیل دیمرهای درونرشتهای پرایمر انتخاب کنید. این عدد را میتوانید با استفاده از «روش نزدیکترین همسایه» (Nearest-Neighbor Method) محاسبه کنید.
- تشکیل دیمر در انتهای $$3^\prime$$ کارایی پرایمر در اتصال به توالی هدف را کاهش میدهد.
- افزایش توالیهای مکمل احتمال هیبریداسیون اشتباه پرایمر را افزایش میدهد.
- تمام بازهای انتهای $$3^\prime$$ پرایمر باید به توالی هدف متصل شود. انتهای $$3^\prime$$ غیر منحصر به فرد احتمال ایجاد جهش اشتباه را افزایش میدهد.
تعیین غلظت یون محلول آزمایش و غلظت پرایمر دو پارامتر دیگری ضروری برای طراحی پرایمر مناسب و محاسبه Tm است. در کادر Salt con غلظت نمک محیط بر اساس میلیمول وارد میشود. مقدار پیشفرض این پارمتر ۳۳ میلیمول و بر اساس محلول سالین سدیم سیترات 2X ۳۳۰ میلیمول در نظر گرفته شده است. در کادر Probe con غلظت پرایمر بر اساس پیکومول وارد میشود. مقدار پیشفرض این پارامتر در نرم افزار ژن رانر ۲۵۰ پیکومول در نظر گرفته شده است. در محیط آزمایشگاه این مقدار با پیشرفت واکنش کاهش مییابد.
طراحی پرایمر توالی یابی در نرم افزار ژن رانر
تعیین دمای ذوب، درصد GC، انتهای $$3^\prime$$ منحصربهفرد و تشکیل نشدن ساختار دوم پارمترهای مهم در طراحی پرایمرهای توالییابی است. الگوریتمهای نرم افزار ژن رانر با بررسی کامل توالی ژنی، تمام پرایمرهای مناسب برای توالییابی با فواصل منظم در را در اختیار کاربر قرار میدهد. برای مثال توالییابی یک DNA با ۲۰۰۰ جفتباز به تعدادی پرایمر در فاصلههای ۲۰۰ تا ۳۰۰ بازی نیاز دارد. الگوریتمهای ژن رانر ابتدا تمام پرایمرهای مطلوب برای ۵۰ باز اول را تعیین میکند. سپس ۲۰۰ باز را انتخاب کرده و پرایمرهای مناسب ۵۰ باز بعدی در دو طرف باز ۲۰۰ام را در اختیار کاربر قرار میدهد و این فرایند تا رسیدن به آخرین باز ادامه مییابد. از این پرایمرها میتوان برای هیبریداسیون قطعه تعیین شده نیز استفاده کرد.
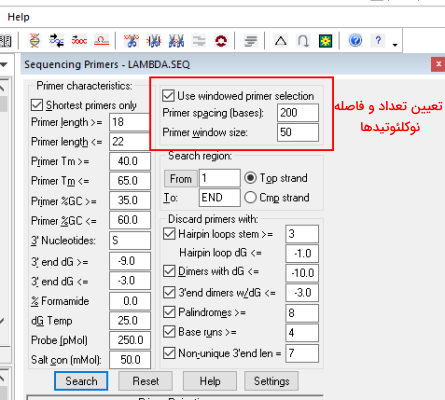
پارامترهای پنل طراحی این پرایمرها شبیه پرایمرهای جهش ژن است. تنها پارامتر متفاوت این پنل تعیین فاصله بین پرایمرها است. با انتخاب این گزینه پرایمرها با فاصله (Primer spacing) و تعداد نوکلئوتیدهای هر طرف باز (Primer window size) مشخص طراحی میشوند. در غیر این صورت پرایمرها بر اساس آنالیز خودکار کل توالی تعیین میشود.
نحوه طراحی پرایمر PCR در نرم افزار ژن رانر چطور انجام می شود؟
واکنش زنجیره پلیمراز (RCR) روشی برای تکثیر تعداد زیادی مولکول DNA از نمونه کم DNA یا RNA است. انجام این روش به دو دسته پرایمر (جلو و معکوس) برای شروع همانندسازی نیاز دارد. نرم افزار ژن رانر بر اساس توالی هدف تمام پرایمرهای مناسب را طراحی میکند. برای استفاده از این امکان نرمافزار از کلید میانبر Ctrl + Shift + P یا مسیر Analysise > Nucleic Acid > PCR primers استفاده کنید.
در پنل این آنالیز میتوانید حدکثر و حداقل طول، درصد GC و دمای ذوب، طول پروب، طول انتهای $$3^\prime$$، غلظت پروب، نوع نوکلئوتیدهای انتهای $$3^\prime$$ و ویژگیهای نامطلوب پرایمر را تعیین کنید. یکی از تفاوتهای پنل طراحی پرایمر PCR با پرایمرهای دیگر امکان اضافه کردن توالی ویژه به انتهای $$5^\prime$$ با استفاده از گزینههای "Sense 5 ext"، "A-sense 5 ext" و "RE sequence" است. در کادر این گزینهها میتوانید توالی نوکلئوتیدی پروموتر (Sense 5 ext و A-sense 5 ext) یا جایگاه شناسایی آنزیم محدودکننده (RE sequence) مورد نظر خود را به انتهای $$5^\prime$$ پرایمر اضافه کنید.
برای طراحی پرایمرهای بهینه الگوریتم ژن رانر اولین باز مشخص شده برای انتهای $$3^\prime$$ در پنل آنالیز را در توالی ژنی پیدا کرده و به اندازه طول پرایمر تعیین شده بازهای بعدی را میخواند. سپس با توجه به پارامترهای تعیین شده تمام پرایمرهای احتمالی را در اختیار کاربر قرار میدهد. این فرایند در تمام طول توالی ژنی تکرار میشود.
پس از انجام آنالیز در بخش پایینی پنل تعداد کل پرایمرهای احتمالی، پرایمرهایی که با ویژگیهای موردنظر همخوانی ندارند و پرایمرهای مطلوب بر اساس فیلترهایی تعیین شده مشخص میشود.
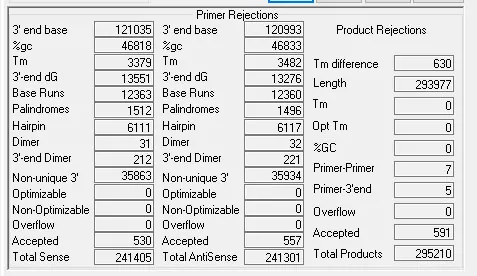
در پنجره سمت چپ طول پرایمر، دمای ذوب، توالی نوکلئوتیدی، شماره باز انتهای $$5^\prime$$ پرایمر در توالی هدف (Strat)، شماره باز انتهای $$3^\prime$$ در توالی هدف (Stop) و درصد GC مشخص میشود.
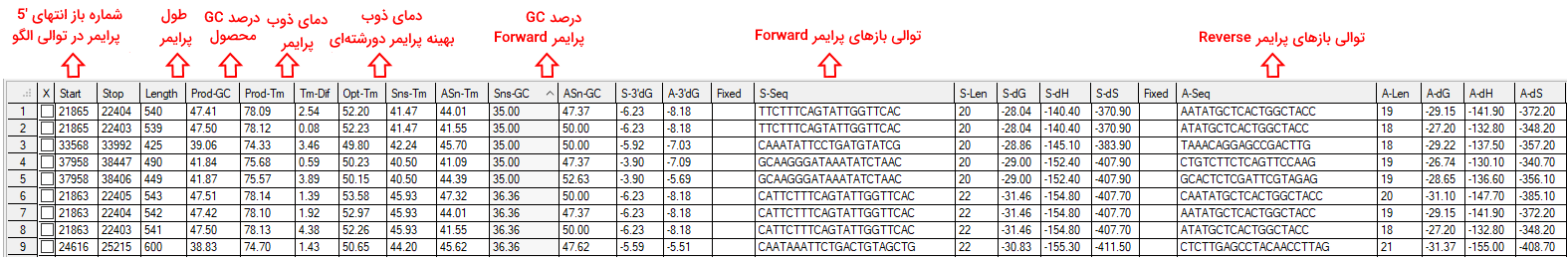
اصلاح پرایمر در ژن رانر
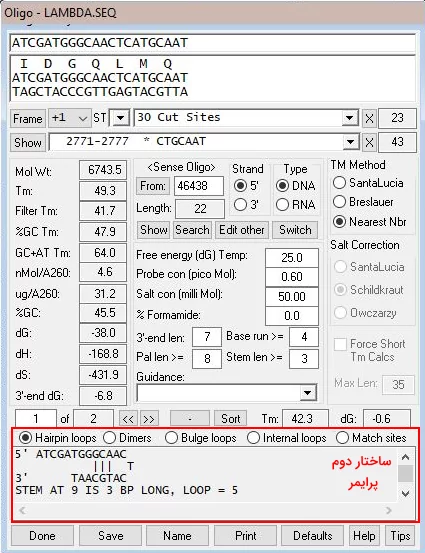
نرمافزار ژن رانر این امکان را فراهم میکند که بهینگی پرایمری که از روشهای دیگر طراحی شدهاند را بررسی کنید. برای استفاده از این امکان از کلید میانبر Ctrl + L یا Analysise > Oligo Analysis مسیر استفاده کنید. الگوریتمها این بخش دما ذوب، درصد GC، ساختارهای دوم و دیگر ویژگیهای مهم پرایمر بهینه را نشان میدهد.
محاسبه دمای ذوب
یکی دیگر از امکاناتی که ژن رانر در اختیار کاربران قرار میدهد، محاسبه سریع دمای ذوب توالیهای طولانی است. برای استفاده از این امکان توالی DNA یا RNA مورد نظر را در پنجره فعال باز کرده و سپس از بخش آنالیز در نوار ابزار گزینه "Melting Temperature" را انتخاب کنید.پنجره باز شده از بخشهای Parameter، Region و Result تشکیل شده است.
- Parameter: در این بخش درصد جفتبازهای غیرمکمل (پیشفرض 0)، غلظت یونهای محیط و درصد فورامید محلول آزمایش را وارد کنید.
- Region: در این بخش شماره باز شروع آنالیز (From) و باز پایانی (To) را مشخص کنید.
- Result: در این بخش نرمافزار بر اساس اطلاعات وارد شده و فرمول «بالدینو» (Baldino)، دمای ذوب و درصد CG توالی را در اختیار شما قرار میدهد.
آنالیز پروتئین در نرم افزار ژن رانر
استفاده از توالی پروتئینی برای طراحی پروبهای اولیگونوکلئوتیدی یکی از روشهای انتهاب توالی کدکننده از کتابخوانههای DNA کلون شده است. ژن رانر این پروبها را با استفاده از ترجمه معکوس توالیهای آمینواسید بهوسیله جدولهای نرمافزار در اختیار کاربر قرار میدهد. برای استفاده از این امکان پس از باز کردن فایل توالی آمینواسیدی در پنجره فعال از کلید میانبر Ctrl + Shift +E یا Analysise > Protein > Probe مسیر استفاده کنید. پنل این آنالیز از بخشهای "Probe length"، "GC content"، "Buffer"، "Search Region"، "Tm" و "Premutations" تشکیل شده است.
- Probe Length: در این بخش تعداد حداقل و حداکثر بازهای موجود در پروب مدنظر خود را تعیین کنید. پروبهای خارج از این محدوده از گزارش نهایی حذف میشوند.
- GC Contetnt: در این بخش حداقل و حداکثر تعداد بازهای C و G در پروبهای مورد نظر را انتخاب کنید.
- Buffer: غلظت یون و فورمامید محلول آزمایش را برای محاسبه Tm در این بخش وارد کنید.
- Search region: در این بخش شماره بازهای متناظر آمینواسید برای شروع و پایان برای بررسی پروبهای مناسب را مشخص کنید.
- Tm: در این بخش حداقل و حداکثر دما ذوب قابل قبول برای پروب را با توجه به شرایط آزمایش مشخص کنید. پروبهایی که در این محدوده نباشند از گزارش نهایی حذف میشوند.
- Premutation: در این بخش میتوان حداقل و حداکثر تعداد تغییرات کدون در پروب را مشخص میکند. مطلوبترین پروب توالی است که بیشترین طول و کمترین تغییرات کدون را داشته باشد.
آنالیز هم ترازی
آنالیزهای همترازی ژنوم برای بررسی رابطه تکاملی ژنوم دو یا چند گونه با استفاده از توالی DNA، RNA یا آمینواسیدی در دسترس انجام میشود. برای استفاده از این امکان نرم افزار ژن رانر از بخش File در نوار ابزار روی گزینه New کلیک کرده و "Alignment Project" را انتخاب کنید. در پنجره باز شده اسم مورد نظر (Name) و توضیحات پروژه (Description) را در کادرهای تعیین شده وارد و نوع توالی در هدف خود را مشخص کنید. با انتخاب کلید OK پنجره این آنالیز باز و گزینههای آنالیز همترازی در بخش آنالیز نوار ابزار فعال میشود.
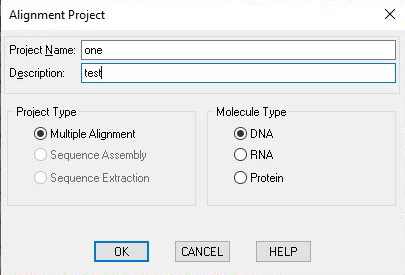
پس از باز شدن پنجره پروژه از بخش Files در نوار پایینی پنجره فابلهای مورد نظر خود را وارد کنید. به این نکته توجه داشته باشید که تمام فایلهای وارد شده باید از یک نوع توالی (پروتئینی یا اسیدنوکلئیک) باشند. با انتخاب کلید Compute Alignment از بخش آنالیز نوار ابزار الگوریتمهای نرمافزار ژن رانر همترازی دو توالی انتخاب شده را محاسبه و بازهای موردنظر را هایلایت میکند.
نحوه آنالیز کلونینگ در نرم افزار ژن رانر چطور انجام می شود؟
کلونینگ یکی از روشهای مولکولی برای تکثیر ژنها در محیط آزمایشگاهی است. در این روش ژن مورد نظر بهوسیله آنزیمهای محدودکننده، نوکلئازها و لیگازها وارد ژنوم یک پروکاریوت یا مخمر میشود. تعیین نوع آنزیم محدودکننده، تعداد جایگاههای برش آنزیم و نوع انتهای ایجاد شده پس از برش قبل از شروع کلونینگ اهمیت زیادی دارد. نرمافزار ژن رانر امکان بررسی این پارامترها را فراهم میکند و برای استفاده از این امکان از کلید میانبر Ctrl + N یا مسیر استفاده کنید.
پنجره باز شده از یک بخش نمایش ویژگیهای توالی حامل (وکتور) و یک بخش ویژگیهای توالی هدف تکثیر (Insertion) تشکیل شده است. با کلیک بر گزینه "Find Sequence" ویژیگیهای این دو توالی را تعیین کنید.
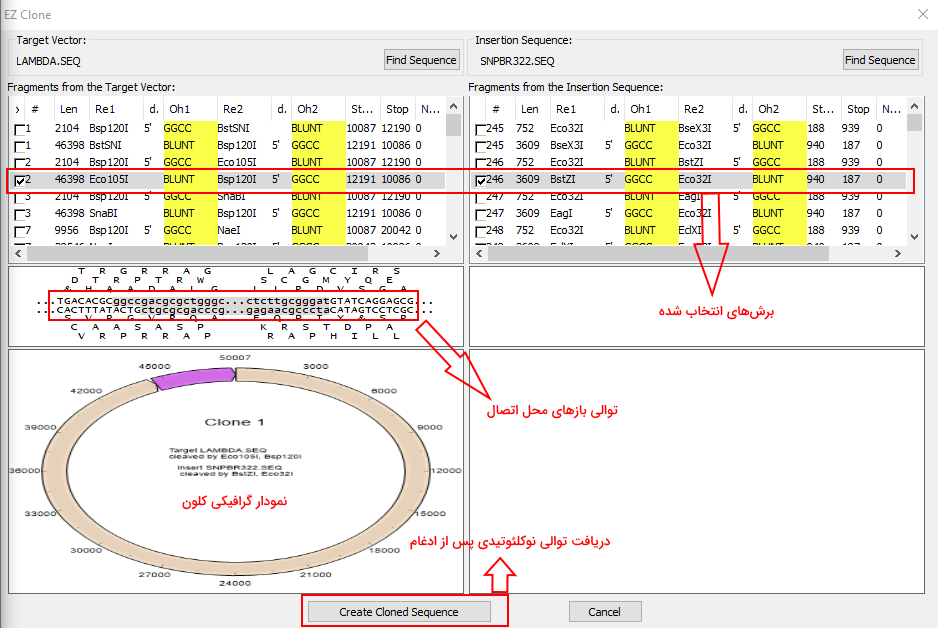
در پنجره بعدی فایل توالی نوکلئوتیدی ژن هدف و وکتور را از فایلهای سیستم خود انتخاب کنید. در بخش "Select Digestion Type" تعداد برش ایجاد شده در دو رشته نوکلئوتیدی و محدوده برش تعیین میشود. با تنظیم این پارامترها میتوانید آنزیمهایی که در جایگاههای تنظیمی وکتور یا ژنهای عملکردی توالی هدف برش ایجاد میکنند را حذف کنید.
در بخش "Select Digestion Constrains" نوع و طول انتهای ایجاد شده پس از برش آنزیمی را تعیین کنید. در بخش آخر فایل جدول آنزیمهای محدودکننده سیستم یا فایل جدول مورد نظر خود را وارد کرده و پس کلیک بر گزینه OK نوع آنزیم، تعداد و جایگاه برش ایجاد شده در دو DNA را دریافت کنید. با انتخاب برش ایجاد شده در دو جدول نرمافزار شکل گرافیکی کلون ایجاد شده و جفتبازهای نزدیک آن را در اختیار شما قرار میدهد. با کلیک بر گزینه "Creat Clone Sequence" توالی نوکلئوتیدی وکتور همراه با ژن هدف را مشاهده میکنید.
جمعبندی
در این مطلب از مجله فرادرس توضیح دادیم که نرم افزار ژن رانر یکی از نرمافزارهای آفلاین و رایگان برای طراحی پرایمرهای RCR، توالییابی، هیبریداسیون و ایجاد جهش ژنتیکی است. این نرمافزار امکان تعیین توالی نوکلئوتیدی پس از ادغام DNA هدف و وکتور، بررسی همترازی پروتئینها یا اسیدهای نوکلئوئيک و محاسبه دمای ذوب رشتههای نوکلئوئیدی را فراهم میکند.