متیلاسیون DNA چیست؟ – در اپی ژنتیک و به زبان ساده

تمام صفات موجودات زنده بهوسیله توالیهای مشخصی در مولکول DNA به نام ژن از نسلی به نسل دیگر منتقل میشود. DNA از تعداد زیادی نوکلئوتید که با هم پیوند کوالانسی دارند، تشکیل شده است. جایگزینی نوکلئوتید با نوکلئوتید دیگر، حذف یا اضافه شدن نوکلئوتیدها و تغییرات تعداد یا ساختار کروموزومها منجر به ایجاد جهش، تنوع صفات و در برخی از موارد بروز بیماریهای ژنتیکی میشود. این تغییرات به دلیل اختلال در جدا شدن کرومووزمها در میتوز، خطا در مکانیسمهای ترمیم DNA و عوامل محیطی ایجاد میشود. تغییرات اپیژنتیک یکی دیگر از عوامل تنوع صفات است. مکانیسمهای این فرایند بدون ایجاد تغییر در ساختار یا نوع نوکلئوتیدها با اضافه شدن گروههای شیمیایی به DNA یا هیستونها بیان ژن و در نتیجه صفات موجود زنده را تغییر میدهند. متیلاسیون DNA یکی از تغییرات اپیژنتیک است.
اضافه شدن گروههای متیل به DNA و اضافه شدن گروههای استیل، متیل و فسفات به هیستونها تغییراتی ست که بیان ژنها را تغییر میدهد. این گروهها پیوندهای غیر کوالانسی پروتئینها با DNA را تغییر میدهند. در نتیجه DNA فشردهتر یا بازتر میشود. در این مطلب از مجله فرادرس مکانیسم متیلاسیون DNA در پستانداران و باکتریها، و نقش آن در تنظیم بیان ژن را توضیح میدهیم.
متیلاسیون DNA چیست؟
تغییرات اپیژنتیکی بدون ایجاد تغییر توالی نوکلئوتیدهای DNA بیان ژنها را تغییر میدهند. این تغییرات برگشتپذیر ممکن است از نسلی به نسل دیگر منتقل شود. اضافه شدن گروههای متیل یا متیلاسیون DNA یکی از این تغییرات اپیژنتیک در ژنوم پروکاریوتها، گیاهان و جانوران است که به تنظیم بیان ژن کمک میکند. در طول تکامل و رشد جنین انسان الگوی متیلاسیون در ژنها تغییر میکند. به همین دلیل تمام بافتها از ژنوم یکسانی تشکیل شده است، اما گروه مشخصی از ژنها در هر بافت بیان میشود.
مکانیسم متیلاسیون DNA
DNA از دو رشته نوکلئوتیدی تشکیل شده که ساختاری مارپیچی (هلیکس) دارند. هر نوکلئوتید DNA از یک باز پورین یا پیریمیدین، قند دئوکسی ریبوز و سه گروه فسفات تشکیل و بر اساس نوع بازها به انواع آدنوزین تریفسفات (ATP)، سیتوزین تریفسفات (CTP)، گوآنوزین تریفسفات (GTP) و تیمیدین تریفسفات (TTP) تقسیم میشود. گوانین و آدنین بازهای پورین هستند که از دو حلقه و سیتوزین و تیمین بازهای پیریمیدین هستند که از یک حلقه نیتروژنی تشکیل میشوند.
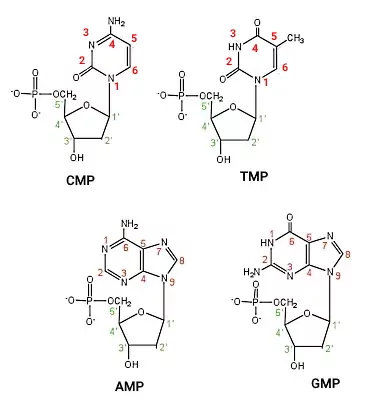
برای تشکیل هر رشته DNA گروه فسفات کربن $$5^\prime$$ قند یک نوکلئوتید با پیوند کواالانسی به گروه OH کربن $$3^\prime$$ قند نوکلئوتید بعدی متصل میشود. در نتیجه در یک انتهای هر شته DNA یک گروه OH و در انتهای دیگر آن یک گروه فسفات آزاد (بدون پیوند) وجود دارد.
گروههای متیل به کربن ۵ نوکلئوتیدهای سیتوزین اضافه میشود. آنزیمهای DNA متیلترانسفراز (Dnmts) گروه متیل S-آدنیل متیونین (SAM) را به سیتوزین اضافه میکند. Dnmt1 در طول همانندسازی DNA گروههای متیل با الگوی رشتههای والدی به رشتههای در حال سنتز اضافه میکند. Dnmt3a و Dnmt3b با ایجاد الگوهای جدید متیلاسیون DNA به تنظیم بیان ژنها کمک میکنند. جایگاه تنظیمی این آنزیمها در انتهای N و جایگاه کاتالیزوری آنها در انتهای C زنجیره پلیپپتیدی قرار دارد. Dnmt3a تقریبا در تمام بافتها میشود.
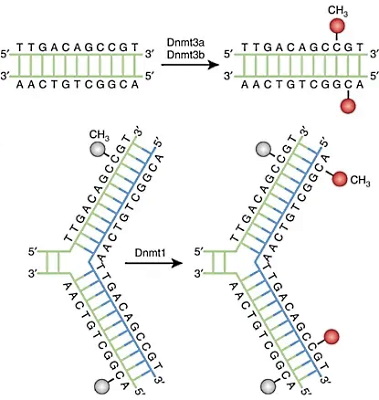
Dnmt3b در سلولهای تمایزیافته به جز سلولهای تیروئید، بیضهها و مغز استخوان بیان نمیشود. Dnmt3L یکی دیگر از اعضای خانواده DNA متیل ترانسفرازها است که جایگاه کاتالیزی در آن وجود ندارد. این پروتئین در مراحل اولیه رشد جنین بیان میشود و فعالیت Dnmt3a و Dnmt3b را تحریک میکند. در سلولهای بزرگسال، Dnmt3L فقط در سلولهای زاینده گامت و غده تیموس بیان میشود. اتصال فاکتورهای رونویسی به پروموتر یا اتصال فاکتورهای تنظیمی به جایگاه مهارکننده ژن، سیگنال فراخوانی متیلترانسفرازها است.
نقش متیلاسیون در تنظیم بیان ژن
پروتئینهای دارای دومینهای MBD، UHRF و انگشت روی به با اتصال به ۵-متیل سیتوزین به تنظیم بیان ژن و متیلاسیون کمک میکند. در پروتئینهای MBD یک دومین اتصالی به متیل CpG و یک دومین مهار رونویسی (TRD) وجود دارد. به جز MBD3 و MBD4 سایر پروتئینهای این خانواده به DNA متصل میشوند. MBD3 به دلیل جهش در دومین اتصالی به متیل سیتوزین، به طور مستقیم به DNA متصل نمیشود و MBD4 در حضور پروتئینهای ترمیم باز تیمین، یوراسیل یا ۵-فلورویوراسیل به DNA متصل میشود. Kaiso به دو ۵-متیل سیتوزین متوالی متصل شده و مهار ژن با متیلاسیون را تحریک میکند.
پروتئینهای UHRF، پروتئینهای شبیه یوبیکوئیتین با دومینهای PHD و انگشت حلقه (Ubiquitin-like, Containing PHD and RING Finger domain | UHRF) هستند. این پروتئینها Dnmt1 را به DNA (به ویژه در همانندسازی) متصل و به حفظ الگوی متیلاسیون کمک میکند. Kaiso، ZBTB4، و ZBTB38 پروتئینهایی هستند که با موتیف انگشت روی به DNA متصل میشود. ZBTB4 و ZBTB38 به دو جایگاه CpG پشت سر هم متصل میشوند و تمایل آنها به توالیهای غیرمتیله بیشتر است.
متیلاسیون DNA و هیستون
هیستونها (H1، H2A، H2B، H3 و H4) پروتئینهای بازی با آمینواسیدهای لیزین و آرژنین فراوان هستند که به فشرده شدن DNA یوکاریوتی در هسته کمک میکنند. هسته این پروتئینها از دو دیمر H2A-H2B و یک تترامر H3-H4 تشکیل شده که ساختار سوم آنها شبیه دو نیم دایره تقریبا مساوی است. موتیفهای مارپیچ-پیچ-مارپیچ (Helix-Turn-Helix) هسته هیستونها با DNA برهمکنش میدهد. انتهای آمینی این پروتئینها خارج از مارپیچهای DNA قرار دارد و دومین تغییرات شیمیایی پس از ترجمه (اضافه شدن گروه متیل، استیل، یوبیکوئینیتین و فسفات) است. این تغییرات با کاهش یا افزایش برهمکنش هیستون-dna در تنظیم بیان ژن و همانندسازی نقش دارند. آنزیمهای Dnmts با آنزیمهای تغییردهنده هیستونها به ویژه آنزیمهای مهار بیان ژن برهمکنش میدهند.
Dnmts1 و Dnmts3a به آنزیم هیستون متیل ترانسفراز (SUV39H1) و هیستون استیلاز متصل میشوند. آنزیمهای هیستون متیل ترانسفراز (HMTs) با اضافه کردن متیل به آمینواسیدهای انتهای آمینی هیستون بیان ژن را مهار میکنند. آنزیمهای داستیلاز (HDACs) با جدا کردن گروههای استیل از هیستونها، برهم کنش DNA-هیستون و فشردگی DNA را افزایش میدهند. تغییرات هیستون الگوی متیلاسیون DNA را نیز تغییر میدهد.
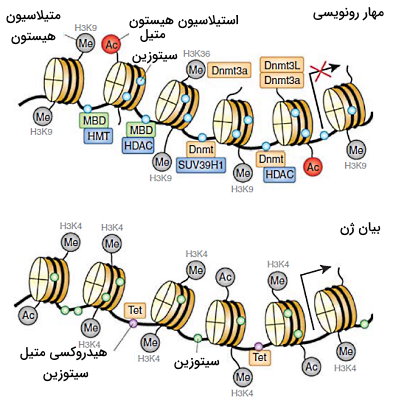
Dnmt3L پس از اتصال به H3 آنزیمهای Dnmt3a و Dnmt3b را به نوکلئوزوم فرا میخواند. اضافه شدن سه گروه متیل به چهارمین لیزین انتهای آمینی (H3K4) این هیستون، اتصال متیل ترانسفرازها و متیلاسیون DNA را مهار میکند. افزایش استیلاسیون هیستونها، دمتیلاسیون DNA را را افزایش می دهد. برهمکنش پروتئینهای MBD و UHRF با هیستونها و DNA به مهار بیان ژن کمک میکند. MeCP2 هیستون داستیلاز و هیستون متیل ترانسفراز را به ژن فرامیخواند جدا شدن گروههای استیل و اضافه شدن گروههای متیل به نهمین لیزین انتهای آمینی هیستون ۳ (H3K9) بیان ژن را مهار میکند.
متیلاسیون DNA پروکاریوت
هیستونها در DNA پروکاریوتی وجود ندارد. به همین دلیل متیلاسیون DNA نقش بیشتری در تنظیم بیان ژن در این موجودات است. آنزیمهای متیل ترانسفراز پروکاریوتی گروههای متیل S-آدنیل متیونین را به نیتروژن ۶ باز آدنین (6mA)، کربن ۵ باز سیتوزین (5mC) یا گروه آمین کربن ۴ سیتوزین (4mC) منتقل میکند. دومین اتصالی به SAM و کاتالیزی این آنزیمها، توالی آمینواسیدی محافظت شده و با یوکاریوتها مشترک است. اما دومین اتصالی به DNA متفاوتی دارند. متیلاسیون DNA علاوه بر بیان ژن در سیستم دفاعی پروکاریوتها در برابر باکتریوفاژها نقش دارد.
- تنظیم بیان ژن: متیلاسیون آدنوزین با کاهش پایداری ترمودینامیکی، ساختار سهبعدی DNA و اتصال آن به پروتئینها را تغییر میدهد. تغییر اتصال DNA-پروتئین بیان ژنها را تغییر میدهد. «تنوع فاز» (Phase Variation) مکانیسمی است که در پاسخ به تغییرات محیطی و بدون ایجاد جهش، به ایجاد تنوع ژنتیکی باکتری کمک میکند. ژنهای کدکننده مولکولهای ادهسین، پیلی، گیرندههای آهن، لیپوپلیساکاریدها و متیل ترانسفرازها بهوسیله این مکانیسم تنظیم میشود. متیلاسیون توالیهای تنظیمی ژن ها اتصال RNA پلیمراز به DNA را مهار و فاصله زمانی بین حالت فعال و غیرفعال ژن را تغییر میدهد.
- سیستم دفاعی: سیستم دفاعی باکتریها از آنزیمهای متیل ترانسفرازها و آنزیمهای محدودکننده تشکیل شده است. متیلترانسفراز الگوی متیلاسیون DNA را حفظ و آنزیمهای محدودکننده DNA خارجی فاقد این الگو را شناسایی میکنند. آنزیمهای محدودکننده DNA غیرمتیله باکتریوفاژ را برش میدهند و آنزیمهای متیل ترانسفراز گروههای متیل را به توالی شناسایی آنزیم محدودکننده روی DNA باکتری اضافه میکنند. سیستم دفاعی باکتریها بر اساس ساختار زیرواحدها، جایگاه برش، توالی شناسایی DNA و فاکتورهای کمکی آنزیمها به چهار گروه تقسیم میشوند.
- سیستم نوع I از دو زیرواحد متیل تراسفراز، رو زیرواحد آنزیم محدودکننده و یک جایگاه اتصال به DNA تشکیل شده است. آنزیم محدودکننده این سیستم DNA دورشتهای را در فاصله زیادی از جایگاه شناسایی برش میدهد و متیل ترانسفراز گروههای متیل را به دو رشته DNA اضافه میکند.
- سیستم نوع II از دو پروتئین Mod (متیل ترانسفراز) و Res (آنزیم محدودکننده) تشکیل شده است که جایگاه شناسایی DNA مستقل از هم دارند.
- سیستم نوع III کمپلکسی از دو پروتئین Mod و یک پروتئین Res است. Mod گروههای متیل را به یکی از رشتههای DNA در توالیهای غیر پالیندرومیک اضافه میکند و کمپلکس Res1Mod2 مولکول DNA را در فاصله ۲۰ تا ۳۰ جفتبازی برش میدهد.
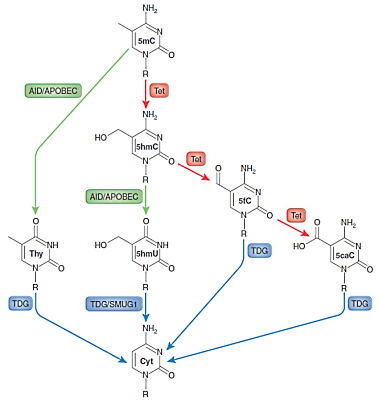
دمتیلاسیون DNA
جدا شدن گروههای متیل از DNA یا دمتیلاسیون به دو روش فعال (با مصرف انرژی) و غیرفعال (بدون مصرف انرژی) انجام میشود. دمتیلاسیون غیرفعال در سلولهای در حال تقسیم انجام میشود. در این سلولها مهار یا اختلال در عملکرد آنزیم Dnmts متیلاسیون DNA دختری را کاهش میدهد. در نتیجه در پایان هر میتوز متیل سیتوزینهای کمتری در ژنوم سلول وجود دارد.
دمتیلاسیون فعال در سلولهای در حال تقسیم و سلولهایی که تقسیم نمیشوند، انجام میشود. در این فرایند چند مرحله واکنشهای اکسایس-کاهش و دآمیناسیون متیل سیتوزین را به نوکلئوتیدی تبدیل میکند که بهوسیله پروتئینهای مسیر ترمیم جدا کردن باز (BER) شناسایی و بهوسیله آنزیم DNA گلیکوزیداز تیمین (TDG)، با سیتوزین غیرمتیله جایگزین میشود. گروه متیل و آمین دو جایگاه فعال متیل سیتوزین در واکنشهای شیمیایی هستند که در مکانیسمهای مختلفی تغییر میکنند.
- دآمیناسیون متیل سیتوزین را بهوسیله کمپلکس سیتوزین دآمیناز القاشده و کمپلکس آنزیم ویرایش mRNA آپوپروتئین بی (AID/APOBEC)، به تیمین تبدیل میکند. پروتئینهای مسیر BER ناجور باز G-T را شناسایی و نوکلئوتید T را با C جایگزین میکنند.
- آنزیمهای ترانسلوکاز ده-یازده (Tet1، Tet2 و Tet3) یک گروه هیدروکسیل به ۵-متیل سیتوزین اضافه میکند. دو مسیر برای تغییر ۵-متیل هیدروکسیل سیتوزین در پستانداران وجود دارد. در یکی از این مسیرها متیل هیدروکسیل سیتوزین با چند مرحله اکسیداسیون اول به ۵-فرمیل سیتوزین و سپی به ۵-کربوکسی سیتوزین تبدیل میشود. در مسیر دیگر ۵-متیل هیدروکسیل بهوسیله کمپلکس آنزیمی AID/APOBEC به ۵-هیدروکسی متیل یوراسیل تبدیل میشود.
نقش متیلاسیون DNA در سرطان
متیلاسیون DNA یکی از تغییرات اپیژنتیکی است که به کنترل بیان ژنهای مختلف ازجمله پروتئینهای کنترل چرخه سلولی کمک میکند و سرطان بیماریای است که به دلیل اختلال در کنترل چرخه سلولی ایجاد میشود. افزایش (هایپرمتیلاسیون) و کاهش (هیپومتاسیون) متیلاسیون ژنها در ایجاد و پیشرفت سرطان نقش دارد. متیلاسیون DNA یکی از مکانیسمهای تنظیمی ژنهای مهارکننده تومور است. افزایش متیلاسیون در پروموتر این ژنها منجر به خاموش شدن این ژنها و تقسیم کنترل نشده سلولها میشود. به علاوه ژنهای تنظیمکننده چرخه سلولی و ترمیم DNA در بسیاری از سلولهای سرطانی بهوسیله افزایش متیلاسیون مهار میشود. BRCA1 یکی از ژنهای ترمیم DNA است که بهوسیله هایپرمتیلاسیون در تومورهای پستان و تخمدان مهار میشود.
O-متیل گوانین متیل ترانسفراز یکی از آنزیمهای ترمیم آلکیلاسیون DNA است. هایپرمتیلاسیون پروموتر ژن این آنزیم احتمال ایجاد جهش در ژن پروتئینهای p53 و K-Ras را افزایش میدهد. پروتئین p53 با القای آپوپتوز در سلولهایی با DNA جهشیافته، تقسیم سلول را مهار میکند. K-Ras پروتئین G مسیر انتقال پیام MAP کیناز است که در انتقال پیام فاکتورهای رشد نقش دارد. MLH1 یکی از پروتئینهای مسیر ترمیم ناجور بازها است که هایپرمتیلاسیون پروموتر آن، احتمال ابتلا به بسیاری از تومورها ازجمله تورمورهای کلونورکتال و اندومتریوم رحم را افزایش میدهد.

miRNA، snoRNA و lncRNA مولکولهای RNA غیرکدشونده هستند که در تنظیم بیان ژنهای تقسیم و تمایز سلولها نقش دارند. miRNA اولیه ساختار سنجاقسری دارد. آنزیم دایسر سیتوپلاسم این RNA را به miRNA دورشتهای ۲۲-۲۳ نوکلئوتیدی تبدیل میکند. این مولکول همراه «کمپلکس القای خاموشی ژن» (miRNA-Induced Silencing Complex | miRISC) به mRNAهای سلول متصل و بیان ژن را مهار میکند. snoRNA مولکولهای RNA کوچک در هسته هستند که در اضافه شدن گروههای متیل و سودویوریدیل به سایر RNAها نقش دارند. lncRNA توالیهای بیش از ۲۰۰ نوکلئوتیدی RNA هستند که در تنظیم بیان ژن شرکت میکنند. هایپرمتیلاسیون تصادفی این ژنها، تنظیم بیان ژن را تغییر و احتمال ابتلا به سرطان را افزایش میدهد.
هیپومتیلاسیون ژن در تومورهای پیشرفته و متاستاتیک بیشتر از تومورهای اولیه است. در این فرایند متیلاسیون توالیهای درون ژنی و بین ژنی کاهش مییابد. تعداد توالیهای تکراری و تراسپوزونها در این مناطق بیشتر است که متیلاسیون رونویسی آنها را مهار کرده است. هیپومتیلاسیون این بخش از ژنها منجر به رونویسی mRNAهای غیرطبیعی میشود. L1NE1 (عنصر بلند پراکنده هستهای ۱) یکی از تراسپوزنهایی است که در بیشتر جهشهای ژنوم انسان نقش دارد و با متیلاسیون مهار میشود. هیپومتیلاسیون پروموتر این ژن، پروموتر جایگزین ژن گیرنده فاکتور رشد هپاتوسیتها (MET) را فعال میکند. افزایش بیان ژن MET اندازه رشد تومور، رگزایی و متاستاز را افزایش میدهد.
آنالیز متیلاسیون DNA
در بخشهای قبلی این مطلب از مجله فرادرس توضیح دادیم که متیلاسیون DNA نقش مهمی در تنظیم بیان ژنها دارد. تغییر فراوانی یا محل متیلاسیون DNA در فیزیولوژی بدن اختلال ایجاد میکند. در نتیجه آنالیز این فرایند به تشخیص و تجویز رروش درمانی مناسب کمک میکند. توالییابی بیسولفیت یکی از روشهای متداول بررسی کمی و کیفی متیلاسیون DNA است. در این روش DNA استخرج شده در نمونه با محلول بیسولفیت مخلوط میشود. بیسولفیت سیتوزینهای غیرمتیله DNA تکرشتهای را به یوراسیل تبدیل میکند. در PCR این نوکلئوتید به عنوان تیمین شناسایی میشود. در نتیجه جفتبازهای CG در DNA نمونه به جفتباز AT در DNA حاصل از تبدیل میشود. بیسولفیت با ۵-متیل سیتوزین واکنش نمیدهد. با این روش میتوان سیتوزینهای غیرمتیله را از ۵-متیل سیتوزین تشخیص داد.
غیر فعال شدن کروموزوم X
برخلاف کروموزوم Y ژنهای زیادی روی کرومووزم X وجود دارد. روی هر کروموزوم X حدود ۱۰۰۰ ژن و در هر کروموزوم Y بین ۱۰۰ تا ۳۰۰ ژن قرار دارد. ژنوم جنس ماده پستانداران از دو کروموزوم X تشکیل شده است. غیرفعال شدن یکی از کروموزومهای X در مراحل اولیه تکامل جنین در این سلولها از اختلال در عملکرد بدن به دلیل افزایش بیان ژنها پیشگیری میکند. کروموزوم X پس از غیرفعال شدن به ساختار بسیار فشرده، کوچ و کروی به نام جسم بار تبدیل میشود. این کروموزومها با دو مکانیسم تصادفی و غیرتصادفی با آنزیمها و RNAهای مشترک غیرفعال میشوند. در پستانداران جفتدار ازجمله انسان، غیرفعال فعال شدن کروموزوم x تصادفی است. اما کروموزوم X که غیرفعال شده در تمام طول عمر سلول و در نسلهای بعدی آن غیرفعال باقی میماند.
RNA رونویسی شده از دو ژن XIST و TSIX در کروموزومهای X غیرفعال شدن کروموزوم را تنظیم میکند. این ژنها فقط در سلولهایی بیان میشوند که حداقل دو کروموزوم X وجود دارد. XIST (رونوشت مخصوص کروموزم x غیرفعال | X-Inactive Specific Transcript) یکی از lncRNAها است که با اتصال به رشته DNA که از آن رونویسی شده، بیان ژنها را غیرفعال میکند. اتصال RNA-DNA در این فرایند با جدا شدن گروههای عاملی جداکننده هیستون از DNA، اضافه شدن گروههای فشردهکننده هیستون-DNA، متیلاسیون سیتوزینها و فراخوانی پروتئینهای اتصالی به هتروکروماتین همراه است.
در این فرایند بخش کمی از ژنها (حدود ۱۵ تا ۳۰٪) غیرفعال نمیشود. بعضی از ژنها در کروموزوم X تمام سلولهای بدن فرد فعال و بعضی بر اساس نوع سلول متفاوت هستند. اختلال در فرایند غیرفعال شدن کرومووزم X و ژنهای فعال آن ممکن است به بیماریهای ژنتیکی وابسته به کروموزوم X، بیماریهای وابسته به تغییر تعداد کروموزوم و بیماریهای وابسته به بیان ژنها در دو کروموزوم X منجر شود.
سوالات متداول
در این بخش از مطلب مجله فرادرس تعدادی از سوالات متداول پیرامون دمتیلاسیون DNA و تغییرات اپیژنتیکی را توضیح میدهیم.
نقش تغییرات اپی ژنتیک در بیماری های خودایمنی چیست؟
عملکرد طبیعی سیستم ایمنی بدن به توانایی فعال شدن در برابر آنتیژنهای خارجی و غیرفعال بودن در برابر آنتیژنهای خودی است. بسیاری از فعالیتهای سلولهای ایمنی ازجمله تمایز سلولهای خونساز، بازآرایی ژن گیرندههای آنتیژنی، بیان اللها و ایجاد پاسخ ایمنی در برابر پاتوژنها بهوسیله مکانیسمهای اپیژنتیکی تنظیم میشود. تغییر این مکانیسمها در پاسخ به تغییرات محیطی احتمال ابتلا به بیماریهای خودایمنی را افزایش میدهد. اختلال در غیرفعال شدن کروموزوم X یکی از تغییرات اپیژنتیکی است که با افزایش ابتلا به بیماریهای خودایمنی همراه است. همچنین هیپومتیلاسیون ژن هیستون استیلاز ۱، هایپراستیلاسیون H3 و H4 و هیپومتیلاسیون نهمین لیزین انتهای آمینی HJ3 از تغییرات اپیژنتیکی در بافت مفصلی افراد مبتلا به روماتوئید آرتریت است.
نقش متیلاسیون DNA در درمان بیماری ها چیست؟
بررسی متیلاسیون DNA به تشخیص نوع تومورها، اثربخشی درمان، پیشبینی اثزبخشی درمان و انتخاب روش درمانی مناسب در بیماران مبتلا به سرطان کمک میکند. الگوی متیلاسیون DNA در تومورها تغییر میکند. با مهار آنزیمهای متیل تراسفراز ازجمله ۵-آزا-۲-دئوکسی سیتیدین و «دسیتابین» (Decitabine) که آنالوگهای سیتوزین هستند، میتوان از گسترش تومورها پیشگیری کرد. این ترکیبات در همانندسازی DNA شرکت میکنند. متیل تراسفرازها این ترکیبات را به جای سیتوزین شناسایی میکنند. ساختار متفاوت آنالوگهای سیتوزین اتصال محکم متیلترانسفرازها به DNA را ضعیف و سیستم ترمیم را فعال میکند. در نهایت متیل ترانسفراز بهوسیله پروتئازها تجزیه و متیلاسیون الگوهای توموری متوقف میشود.