طراحی اسکریپت ژنتیک در پایتون — به زبان ساده

در این مقاله در خصوص برخی آرگومانهای خط فرمان و ورودی کاربر صحبت میکنیم که طراحی اسکریپت ژنتیک را در پایتون میسر ساختهاند.
ساخت رابطهای کاربری
در این بخش مراحل ابتدایی برای ساخت یک رابط کاربری با استفاده از ورودی تعاملی کاربر و آرگومانهای خط فرمان را مورد بررسی قرار میدهیم. کدی که در این مقاله ارائه شده است، در طی بخشهای بعدی این سری مقالات بهبود خواهد یافت.
در هنگام طراحی یک اسکریپت، همواره باید کاربر را در نظر داشت. در اغلب موارد، زمانی که یک اسکریپت را برای حل یک مشکل مینویسیم، این احتمال وجود دارد که مشکل افراد دیگری را نیز حل کنیم. با در نظر داشتن این ذهنیت همواره باید میزان سهولت استفاده از برنامه خودتان از سوی افراد دیگر را نیز مورد توجه قرار دهید. ما در این مقاله با پیروی از یک سری قواعد کلی مثالهایی از اسکریپتهای ژنتیک ارائه میکنیم، اما ایدههای مطرح شده در آن میتوانند به صورت گستردهای برای طراحی هر برنامه دیگری نیز استفاده شوند.
سناریوی زیر را در نظر بگیرید. ما میخواهیم یک توالی DNA را مورد جستجو قرار دهیم و تعیین کنیم که آیا دو جایگاه آنزیم محدودکننده در بالاتر از یک آستانه تعیینشده وجود دارند یا نه. برای نمونه آیا توالی آنزیمهای محدودکننده برای EcoRI و HindIII در یک توالی DNA بیش از 5، 10 یا 15 بار حضور دارند؟
بدین ترتیب برنامه پایتون ما باید شرایط زیر را داشته باشد:
- انعطافپذیر باشد: یعنی کاربر امکان جستجوی جایگاههای محدودکننده یا در واقع هر موتیف DNA در یک توالی DAN را دارد و تعداد تکرار آن را بالاتر از یک حد آستانه مشخص میسازد.
- امکان کنترل داشته باشد: هر توالی DNA که کاربر انتخاب میکند را جستجو کند.
- بخشنده باشد: برنامه میبایست اشتباههای معمول کاربران را در نظر بگیرد. برای مثال تایپ کردن نام بازهای DNA بدون استفاده از کاراکترهای ambiguity در یک توالی DNA یا استفاده از حروف کوچک برای بازهای DNA.
- «بسیار» بخشنده باشد: برنامه باید همه تلاش خود را در راستای کمک به کاربر در جهت وارد کردن یک توالی معتبر DNA ارائه کند.
ورودی تعاملی کاربر
برای دریافت ورودی کاربر در یک برنامه باید از تابع ()input استفاده کنیم. تابع ()input یک آرگومان منفرد رشتهای میگیرد که از طریق اعلان نمایش یافته برای کاربر دریافت میشود و مقدار وارد شده را به صورت یک رشته بازمیگرداند. در این حالت بهتر است پیامی به کاربر نمایش داده شود که به صورت روشنی از وی بخواهد مقدار مورد نظر خود را وارد نماید.
در مثال زیر از کاربر میخواهیم که یک توالی DNA را وارد کند:
1DNA = input('Please enter your DNA sequence...')با این وجود، بروز خطا ناگزیر است و یک ضربه نادرست روی کیبورد و یا غلط املایی هر زمان محتمل است. در چنین وضعیتی دو گزینه در اختیار داریم؛ میتوانیم به سادگی از برنامه خارج شویم و همه کارها را از نو آغاز کنیم. برای خروج بیدرنگ میتوانیم کد زیر را اجرا کنیم:
1import re
2DNA = input('Please enter your DNA sequence...').upper()
3while re.search('[^AGCT]', DNA):
4 exit("DNA sequence entered is not valid")با این حال، این کار به صورت شهودی نادرست به نظر میرسد. به طور جایگزین میتوانیم امکان وارد کردن مقدار دیگری را در اختیار کاربر قرار دهیم و یا این کار را تا هر تعداد دفعاتی که کاربر یک توالی DNA قابل قبول وارد نکرده است، تکرار کنیم. این کار به نام اعتبارسنجی ورودی کاربر شناخته میشود و اساساً به معنی بررسی معنیدار بودن ورودی ارائه شده از سوی کاربر است. در این مثال، ما میخواهیم که کاربر یک توالی را بدون بازهای مبهم وارد کند. ما میتوانیم از یک گروه کاراکتر منفی شده و تابع ()re.search برای اطمینان یافتن از این که کاربر یک توالی معتبر DNA وارد کرده است استفاده کنیم. همچنین از متد ()upper. استفاده میکنیم تا مطمئن شویم که همه حروف توالی به صورت حروف بزرگ لاتین هستند. این مباحث همگی در حوزه «عبارتهای منظم» (Regular Expressions) قرار میگیرند.
1import re
2DNA = input('Please enter your DNA sequence...').upper()
3while re.search('[^AGCT]', DNA):
4 DNA = input('Please enter a valid DNA sequence...').upper()زمانی که برنامه ورودی کاربر را دریافت کند، باید به دقت در مورد طراحی آن تأمل کنیم. در برخی موارد کاربر یک برنامه را اجرا میکند و این برنامه نیازمند نوعی ورودی کاربر است که این مسئله به طور روشنی مشخص نشده است. ما باید از چنین موقعیتهایی اجتناب کنیم. در چنین وضعیتی کاربر ممکن است توجه خود را به چیز دیگری معطوف کند و برنامه در اعلان خود منتظر ورودی کاربر باشد و بدین ترتیب هیچ پیشرفتی در روند کار صورت نگیرد.
گرچه ورودی کاربر میتواند موجب تعاملی و انعطافپذیر شدن برنامه بشود؛ اما اجرای برنامههایی که بخشی از گردش کار آن به صورت نظارتنشده است کار دشوارتری محسوب میشود. به طور ایدهآل بهتر است موقعیتهایی را طراحی کنیم که برنامه ما همه آرگومانهای تعیینشده را در آغاز از کاربر دریافت کند. برای نیل به این مقصود؛ آرگومانهای خط فرمان یک راهحل ایدهآل محسوب میشوند.
آرگومانهای خط فرمان
آرگومانهای خط فرمان رشتههایی هستند که پس از نام برنامهای که میخواهیم اجرا شود، در خط فرمان تایپ میکنیم. برای استفاده از آرگومانهای خط فرمان در اسکریپتهای پایتون لازم است که ماژول sys ایمپورت شود. سپس میتوانیم با استفاده از لیست خاص بازگشتی از سوی sys.argv به آرگومانهای خط فرمان دسترسی داشته باشیم. عنصر نخست sys.argv، نام برنامه است که در این مورد به صورت Command_line_arguments.py و در اندیس 0 لیست قرار دارد. مثال زیر، اندیسها آرگومانهای متناظر خط فرمان آن را نمایش میدهد.
ما میتوانیم با ایجاد امکان وارد کردن توالی به صورت حروف کوچک یا بزرگ در خط فرمان، کد خود را بیش از این پایدار بسازیم. همچنین میتوان از متد رشته ()upper. به منظور مدیریت این وضعیت بهره جست. با این وجود، یک نکته مهم که باید توجه داشت این است که گزینههای خط فرمان همواره به صورت رشته بازگشت مییابند و از این رو باید count_no را به شکل زیر صریحاً به صورت عدد صحیح تبدیل کنیم:

1import sys
2pattern = sys.argv[1].upper()
3pattern2 = sys.argv[2].upper()
4count_no = int(sys.argv[3])در خط فرمان زیر، به نکاتی که در ادامه آمده است میتوان اشاره کرد:
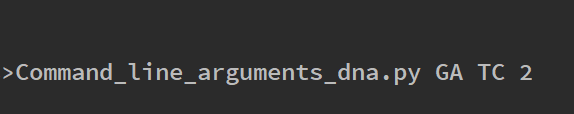
- GA نخستین آرگومان خط فرمان در اندیس 1 است که به الگو اشاره میکند.
- TC دومین آرگومان خط فرمان در اندیس 2 است که به الگوی 2 اشاره میکند.
- 2 سومین آرگومان خط فرمان است که در اندیس 3 قرار دارد و به count_no اشاره میکند.
اسکریپت پایتون ما یک تابع برای جستجوی دو آنزیم محدودکننده درون توالی DNA تعیین شده تعریف میکند. سپس یک دیکشنری میسازیم و در آن به ذخیرهسازی توالی آنزیم محدودکننده به صورت کلید میپردازیم و مقادیر آن نیز شمار آنها خواهد بود. در ادامه روی این دیکشنری یک حلقه تعریف و بررسی میکنیم که آیا توالی بزرگتر از تعداد تعیینشده از سوی کاربر است یا نه. کد کامل اسکریپت به صورت زیر است:
1import re
2import sys
3# DNA = input('Please enter your DNA sequence...').upper()
4# while re.search('[^AGCT]', DNA):
5# DNA = input('Please enter a valid DNA sequence...').upper()
6
7DNA = open('E.coli_genome.fasta').read()
8
9pattern = sys.argv[1].upper()
10pattern2 = sys.argv[2].upper()
11count_no = int(sys.argv[3])
12
13def restriction_enzyme_func(pattern, pattern2):
14 result = []
15 for m in re.finditer(pattern, DNA):
16 result.append(m.group())
17
18 for x in re.finditer(pattern2, DNA):
19 result.append(x.group())
20
21 return result
22
23restriction_site_count = {}
24for pat in restriction_enzyme_func(pattern, pattern2):
25 current_count = restriction_site_count.get(pat, 0)
26 new_count = current_count + 1
27 restriction_site_count[pat] = new_count
28
29
30for pattern, count in restriction_site_count.items():
31 if count > count_no:
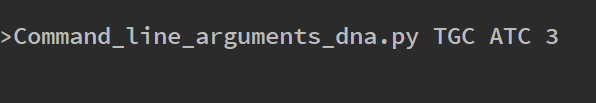
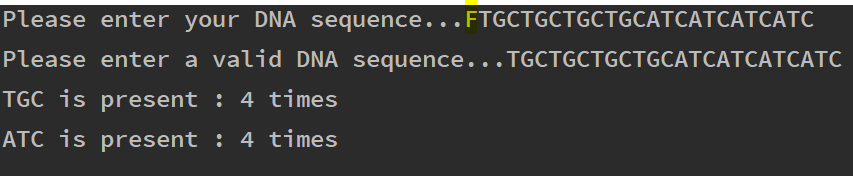
32 print('The restriction enzyme site ' + pattern + ' is present : ' + str(count) + ' times')اکنون میتوانیم اسکریپت را با استفاده از آرگومانهای خط فرمان اجرا کنیم. در این مرحله برای تست کارکرد برنامه از یک توالی کوچک تعیینشده از سوی کاربر استفاده کرده و تلاش میکنیم تعیین کنیم آیا tri-nucleotide-های TGC و ATG بیش از 3 بار وجود دارند یا نه.
در این مثال، میتوانیم به وضوح مشاهده کنیم که کاربر یک نوکلئوتید DNA نامعتبر به صورت F (با رنگ زرد مشخص شده) وارد کرده است. برنامه ما در ادامه از کاربر میخواهد که یک توالی معتبر DNA وارد کند و زمانی که این کار صورت گرفت، نتیجه را بازگشت میدهد.
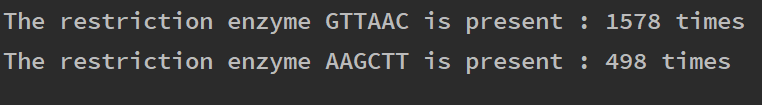
با این حال در اغلب موارد میخواهیم کل ژنوم را مورد جستجو قرار دهیم و از این رو تقاضا برای وارد کردن مقدار از سوی کاربر بهسادگی میسر نیست. برای نمایش بهتر این که این برنامه تا چه حد میتواند مفید باشد ما کل ژنوم E.coli را از NCBI با شماره دسترسی Genbank CU928161.2 دانلود کردیم. این ژنوم نسبتاً بزرگ است و دقیقاً شامل 5،032،268 جفت باز است. با این وجود، اسکریپت ما به طرز کارآمدی آن را مدیریت میکند و نتیجه را تقریباً به صورت آنی بازگشت میدهد. در مثال زیر جستجو کردهایم که جایگاههای آنزیمهای محدودکننده EcoR1 GTTACC, و HindIII AAGCCT در ژنوم بالاتر از 50 بار حضور دارند یا نه. اگر چنین باشد برنامه اعلام میکند که چندین بار این جایگاهها وجود داشتهاند.
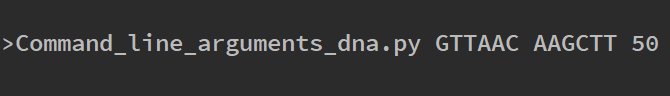
علاوه بر این میتوانیم خروجی بسیار واضحتری از ترمینال بگیریم و بدین ترتیب همه افرادی که نتایج را روی ترمینال میخوانند، میتوانند دادهها را تفسیر کنند.
نتیجهگیری
آرگومانهای خط فرمان روشی مناسب برای دریافت ورودی در برنامههای پایتون محسوب میشود. در این روش همه دادههای متناظر که برنامه نیاز دارد در آغاز ارائه میشوند. در این راهنما مقدمه کوتاهی در مورد روش طراحی اسکریپتهای پایتون با ذهنیت کاربر محور ارائه کردیم. اگر میخواهید کاربر بتواند با برنامه شما تعامل داشته باشد، باید انتظار بروز خطا را نیز داشته باشید و بر همین اساس باید رویههای اصلاح خطا را در اسکریپت خود در نظر بگیرید.
اگر این مطلب برای شما مفید بوده است، آموزشهای زیر نیز به شما پیشنهاد میشوند:
- مجموعه آموزشهای برنامهنویسی پایتون Python
- گنجینه آموزش های برنامه نویسی پایتون (Python)
- مجموعه آموزشهای الگوریتم ژنتیک و محاسبات تکاملی
- زبان برنامه نویسی پایتون (Python) — از صفر تا صد
- آموزش پایتون: ساخت اپلیکیشن دیکشنری — از صفر تا صد
- ترفندهای پایتون که باید آنها را بدانید — راهنمای کاربردی
==












