آموزش نرم افزار Oligo در طراحی پرایمر و آنالیز – به زبان ساده
مولکول DNA از زیرواحدهای نوکلئوتیدی تشکیل شده که در واحدهای ژنیسازماندهی میشوند. بررسی ساختار ژنها، توالیهای نوکلئوتیدی بین آنها و اثر آنزیمهای مختلف بر ژنها کمک فراوانی به تشخیص بیماریها، تولید DNA نوترکیب و تشخیص شباهت DNA بین گونههای مختلف میکند. تکثیر DNA در آزمایشگاه یکی از مراحل اصلی این آنالیزها است. در این فرایند مثل همانندسازیهای داخل سلولی به آنزیمهای پلیمراز و پرایمرها نیاز است. روشهای مختلفی برای طراحی پرایمر وجود دارد. در مطالب قبلی مجله فرادرس طراحی پرایمر با نرمافزار «Gene Runner» را توضیح دادیم. در این مطلب به آموزش نرم افزار Oligo برای طراحی پرایمر میپردازیم.
- میآموزید نرمافزار Oligo چیست و چه کاربردی در طراحی پرایمر دارد.
- یاد میگیرید از نوارهای کاربری Analyze و Search در Oligo استفاده کنید.
- با اجزای پنجره اصلی نرمافزار Oligo و عملکرد آنها آشنا میشوید.
- میتوانید مراحل طراحی و آنالیز پرایمر را با Oligo تشخیص دهید.
- دلایل و کاربرد بررسی ساختار سنجاقسری در طراحی پرایمر را میآموزید.
- نحوه یافتن جایگاه آنزیمهای محدودکننده با توالی پروتئینی را یاد میگیرید.


نرمافزار اولیگو یکی از نرمافزارهای گرافیکی است که به کاربر کمک میکند با چند کلیک ساده پرایمر مورد نیاز برای تکثیر ژنهای مختلف را در اختیار داشته باشد. این نرمافزار با دریافت توالی ژنی و ویژگیهای پرایمر مورد نظر کاربر، تمام پرایمرهای مناسب را طراحی میکند. به علاوه بهوسیله این نرمافزار میتوان دمای ذوب پرایمرهای طراحی شده با روشهای دیگر را بررسی کرد. محاسبه زمان لازم برای هیبریداسیون، آنالیز اتصال پرایمر به جایگاههای غیراختصاصی و آنالیز جایگاه برش آنزیمهای محدودکننده در ژن امکانات دیگری است که نرمافزار Oligo در اختیار کاربر قرار میدهد. یکی از مزیتهای این نرمافزار نوار کاربری ساده و راهنمای کامل سایت آن است. اگر به دنبال آموزش نرم افزار Oligo هستید در این مطلب با ما همراه شوید.
نرم افزار اولیگو چیست؟
طراحی پرایمر یکی از مراحل انجام مطالعات ژنتیکی ازجمله توالییابی ژنها، انجام PCR، سنتز ژن نوترکیب و نشانهگذاری ژنها است. Oligo نرمافزاری برای طراحی و آنالیز پرایمرها است. الگوریتمهای این نرمافزار با استفاده از اطلاعات ترمودینامیک «نزدیکترین همسایه» (Nearest Neighbor) بهترین پرایمر برای توالی مورد نظر را طراحی میکند. این روش از پارامترهای ترمودینامیکی خطی و مستقل جفتبازهای DNA برای پیشبینی تغییرات آنتالپی، آنتروپی و انرژی آزاد گیبس استفاده میکند. علاوه بر طراحی پرایمر به کمک این نرمافزار میتوان جهشهای جهتدار مولکول DNA را بررسی کرد.
لینک دانلود نرمافزار oligo: «+»
آیتمهای مختلف بخش آنالیز این نرمافزار اطلاعات مربوط به ساختار دوم RNA و DNA، تشکیل دیمر، اتصال پرایمر به توالی اشتباه، پایداری داخلی، توالی برش آنزیمهای محدودکننده، ترکیب پرایمر و ویژگیهای فیزیکی آن را در اختیار کاربر قرار میدهد. این نرمافزار برای سیستم عاملهای ویندروز و مک طراحی شده است و کاربر برای استفاده از آن به حداقل ۲۰ مگابایت حافظه داخلی و ۱۲۸ مگابایت RAM نیاز دارد.

آموزش نوار کاربری اولیگو
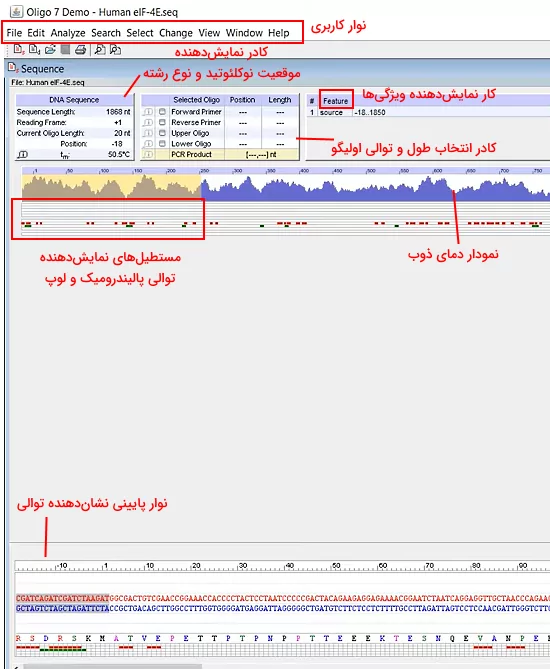
اولین قدم برای آموزش نرم افزار Oligo آشنایی با نوار کاربری نرمافزار است. با کلیک بر آیکون نرمافزار در سیستم، پنجره اصلی برای شما باز میشود. در بالای این پنجره نوار کاربری نرمافزار با گزینههای «File» و «Edit» و «Analyze» و «Search» و «Select» و «Change» و «View» و «Windows» و «Help» وجود دارد. گزینه File امکان بارگذاری فایل در نرمافزار، ذخیره فایل خروجی و پرینت دادهها را در اختیار کاربر قرار میدهد. با کلیک بر گزینه ادیت نوار کشویی برای کاربر باز میشود که که امکان اعمال تغییرات در توالی پرایمر (Forward Primer و Reverse Primer)، کل ژن (Entire Gene) و توالی اولیگومری مورد نظر (Lower Oligo و Upper Oligo) را تغییر دهید.
گزینه Analyze نوار کاربری
به کمک آیتمهای گزینه Analyze کاربر میتواند اطلاعات توالی انتخاب شده (Duplex Info)، تشکیل ساختار دورشتهای در پرایمر (Duplex Formation)، احتمال تشکیل ساختار سنجاقسری (Hairpin Formation)، توالی نوکلئوتیدی پرایمر و دمای ذوب آن (Composition & Tm)، توالیهای مشابهی که ممکن است به پرایمر متصل شود (False Priming Site)، پایداری (Internal Stability)، فراوانی توالی مورد نظر در کل ژنوم (Sequence Frequency)، توالی برش آنزیم محدودکننده در ژن (Restriction Enzyme Site)، توالی برش آنزیم محدود کننده در پروتئین (Restriction Enzyme Site in Protein)، زمان هیبریداسیون (Hybridization Time) و توالی خوانش باز ژن (Open Reading Frame) را بررسی کند.
گزینه Search نوار کاربری
گزینه Search امکان جستجوی پرایمر یا توالی مورد نظر بین بخشهای دیگر ژنوم و توالی برش آنزیمهای محدوکننده را فراهم میکند. گزینه Select امکان انتخاب پرایمرها یا توالی اولیگومری مورد نظر را در اختیار کاربر قرار میدهد. با استفاده از گزینه Change میتوانید قبل از شروع آنالیز پرایمر میتوانید پارامترهای مورد نطر خود را تغییر دهید. با انتخاب View کاربر انتخاب میکند نوار ابزار (Tool Bar) و وضعیت (Status Bar) نمایش داده یا پنهان شود.
آیتم های پنجره اصلی
نوار پایینی پنجره اصلی توالی بارگذاری شده در نرمافزار را نشان میدهد. با کلیک بر هر نوکلئوتید این توالی جایگاه نوکلئوتید و نقطه ذوب آن در دو کادر سمت راست بالای توالی با رنگ قرمز نمایش داده میشود. زیر این نوار ترجمه کدونها با نماد تکحرفی آمینواسید نمایش داده شده است.
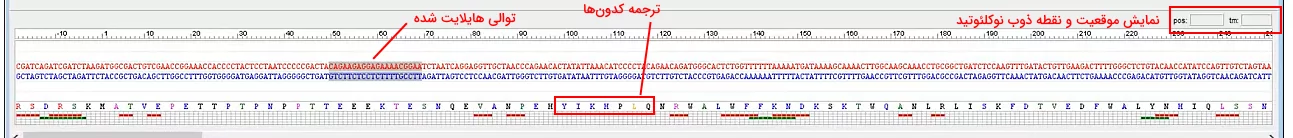
سمت چپ پنجره اصلی زیر نوار کاربری پنجره DNA Sequence قرار دارد. این پنجره طول توالی انتخابی را نشان میدهد. نرمافزرا به شکل پیشفرض ۲۰ نوکئوتید را انتخاب میکند که کاربر میتواند با استفاده از گزینه Change این مقدار را تغییر دهد. نوع رشته ژنوم در بخش Reading Frame مشخص شده است. (۱+) رشته کدشونده و (۱-) رشته مکمل را نشان میدهد. این توالی در نوار پایین پنجره هایلایت شده است. Position شماره اولین نوکلئوتید توالی انتخابی در سمت چپ را نشان میدهد.
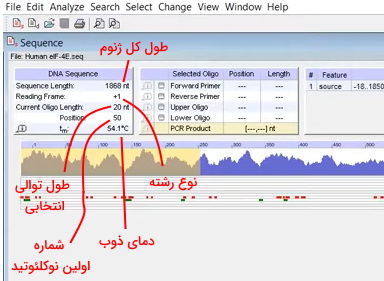
سمت راست DNA Sequence پنجره Selection Oligo قرار دارد. کاربر با استفاده از این پنجره میتواند طول و موقعیت پرایمرهای Forward و Reverse را تعیین کند. «Feature» کادر کنار این پنجره است که ویژگیهای توالی بارگذاری شده در نرمافزار را نمایش میدهد. به طور پیشفرض تعداد نوکلئوتیدهای توالی بارگذاری شده در این بخش نمایش داده میشود. اما کاربر میتواند مشخصات توالی مورد نظر خود را در این کادر وارد کند. نمودار پایین این کادرها، دمای ذوب کل توالی بارگذاری شده را نشان میدهد. با کلیک روی بخشهای مختلف این نمودار توالی انتخابی در نمودار پایین صفحه هایلایت میشود.
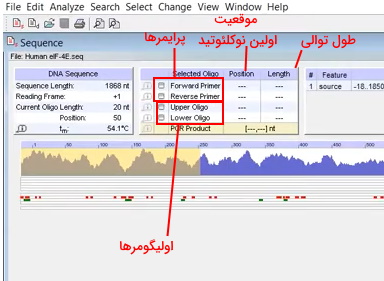
سه مستطیل زیر نمودار دمای ذوب قرار دارد. مستطیل اول به پنج و مستطیل دوم به چهار بخش تقسیم شده است و مستطیل آخر یکبخشی است. بخشهای مستطیل اول به ترتیب محل شروع پرایمر Forward، محل Upper Oligo، توالی بین پرایمرهای Forward و Reverse، محل Lower Oligo و محل پرایمر Reverse را نشان میدهد. بخش اول مستطیل دوم توالیهایی که احتمال تشکیل ساختارهای سنجاقسری پرایمر و DNA زیاد است را با رنگ قرمز مشخص میکند. این توالیها برای طراحی پرایمر مناسب نیستند. بخش دوم توالیهایی که احتمال تشکیل ساختارهای سنجاقسری ضعیف را نشان میدهد. میتوان از این توالیها برای طراحی پرایمر استفاده کرد. اما به این نکته توجه کنید که در دماهای بالا این ساختارها تشکیل نشود. بخش سوم توالیهای پالیندرومیک را با رنگ سبز نشان میدهد و در بخش انتهایی نتیجه جستجوی یک توالی مشخص DNA با رنگ زرد نمایش داده میشود. در مستطیل انتهایی توالی انتخابی از کادر Feature با رنگ بنفش نمایش داده میشود.
آموزش طراحی پرایمر با نرم افزار Oligo
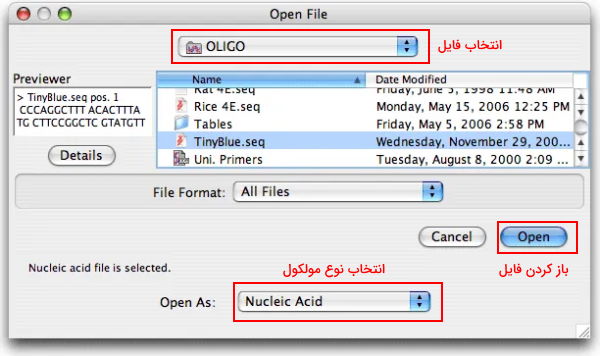
در بخش قبلی آموزش نرم افزار Oligo نوار کاربری نرمافزار را توضیح دادیم. در این بخش قصد داریم با استفاده از نوار کاربری برای توالی مورد نظر خود پرایمر مناسب طراحی کنیم. برای این کار ابتدا از منوی File توالی نوکلئوتیدی را در نرمافزار بارگذاری میکنیم. با انتخاب این گزینه پنجرهای شبیه شکل برای شما باز میشود که امکان انتخاب فایل از سیستم را فراهم میکند. در این پنجره کاربر میتو.اند انتخاب کند توالی نوکلئوتیدی یا ترجمه پروتئینی آن باز شود. این نرمافزار فایلهایی با پسوند «Seq.» و «txt.» را باز میکند.
کاربر میتواند به کمک دابل کلیک در کادر Feature یا انتخاب این گزینه در منوی Edit توالی که به پرایمر آن نیاز دارد را انتخاب کند.با کلیک بر علامت (+) در پایین سمت راست پنجره باز شده ردیف جدیدی برای اضافه کردن توالی مورد نظر کاربر باز میشود. در مستطیل اول نام توالی انتخابی و در مستطیل دوم شماره اولین و آخرین نوکلئوتید توالی به شکل (اولین نوکلئوتید .. آخرین نوکلئوتید) وارد میشود. با استفاده از دستور join در این قسمت کاربر میتوان همزمان دو توالی را برای طراحی پرایمر انتخاب کند. برای مثال عبارت «join(100..400,700..950)» نرمافزار پرایمر را با در نظر گرفتن توالی بین صدمین تا چهارصدمین و هفصدمین تا نهصدمین نوکلئوتید ژنوم طراحی میکند. با کلیک روی گزینه «Accept» پایین و سمت راست پنجره توالی انتخابی شده ذخیره میشود.
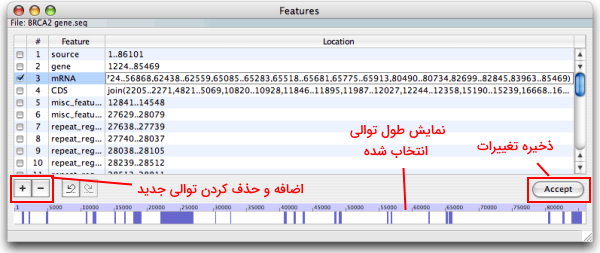
برای طراحی پرایمر میتوان از دو روش استفاده کرد. در روش اول کاربر پرایمر را انتخاب میکند. برای این کار فرض کنید پرایمری با دمای ذوب ۵۳ مدنظر کاربر است. برای پیدا کردن توالی پرایمر با حرکت دادن موس روی نمودار دمای ذوب نقطه شروعی که دمای ذوب آن ۵۳ است را انتخاب میکنیم. سپس در کادر Selection پرایمر Forward یا Reverse را تیک میزنیم. پس از انتخاب این گزینهها محل پرایمر و توالی بین آن در مستطیلهای پایین نمودار دمای ذوب نمایش داده میشود.
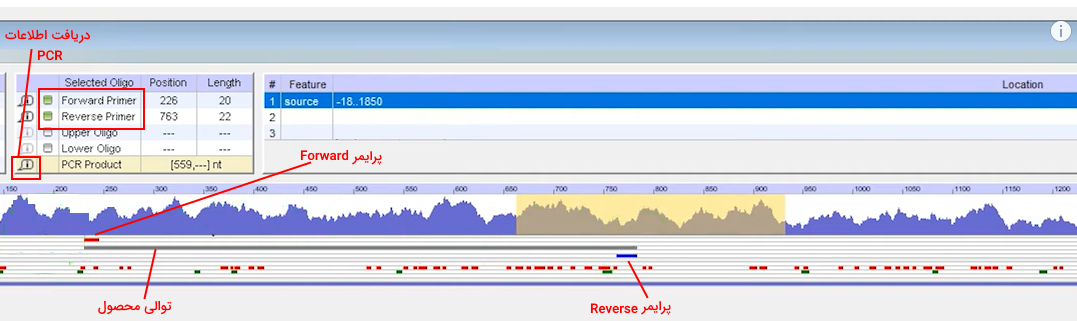
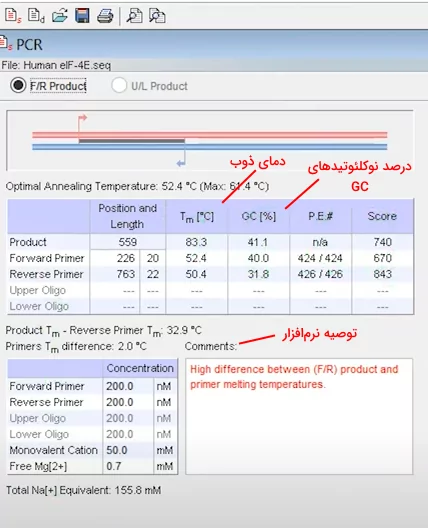
با کلیک بر گزینه PCR دمای ذوب، طول توالی و درصد نوکلئوتیدهای GC پرایمرها و توالی بین آنها نمایش داده میشود. به علاوه نرمافزاز توصیههایی برای طراحی پرایمر بهتر در اختیار کابر قرار میدهد. برای مثال در نمونه زیر اختلاف دمای ذوب پرایمرها و محصول زیاد است.
در روش دوم ویژگیهایی پرایمر مورد نظر را در اختیار نرمافزار قرار میدهیم و نرمافزار مناسبترین پرایمرها را طراحی میکند. برای طراحی پرایمر از این روش در منوی Search آیتم For Primer & Probes را اننتخاب کنید. در پنجره دو بخش «Search Option» و «Subsearch» وجود دارد. در بخش Search Option میتوانید نور پرایمر برای انواع PCR را انتخاب کنید.
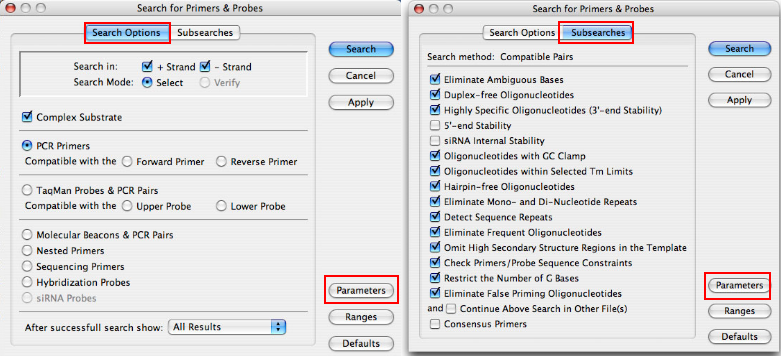
در بخش Subsearch ویژگیهای پارامتر مورد نظر را انتخاب کنید. گزینههای بهینه به صورت پیشفرض در نرمافزار انتخاب شده است. کاربر میتواند با انتخاب گزینه «Parameters» در این پنجره غلظت یونهای تکظرفیتی، یون سدیم و منیزیم، غلظت اسید نوکلئیک و دمای PCR را تغییر دهد. پنجره «Constraints» و «More Constraints» امکان اعمال محدودیتهای بیشتر در طراحی پرایمر را برای کاربر فراهم میکند. با انتخاب گزینه «Range» میتوانید طول قطعه تکثیر شده در PCR و ناحیه نوکلئوتیدی رشته مثبت و منفی را مشخص کنید.
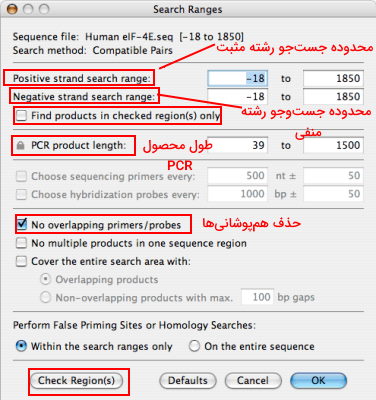
با تیک زدن گزینه «No overlapping primers/probes» پرایمرهایی که همپوشانی دارند از نتایج جستجو حذف میشوند. از گزینه "Check Region" این پنجره میتواند یکی از توالیهایی را انتخاب کنید که قبلا در کادر Feature مشخص کردهاید. بپس از انتخاب توالی مورد نظر گزینه «Find Products in Checked Region(s) Only» در کادر بالای پنجره را تیک بزنید تا نرمافزار تنها توالی انتخاب شده را برای طراحی پرایمر جستوجو کند. در پنجره نتایج شماره اولین نوکلئوتید پرایمرها، Score نرمافزار، طول محصول ، دمای دوباره به هم چسیبدن بهینه (Annealing)، درصد نوکلئوتیدهای GC پرایمرها را نمایش داده میشود. با دابل کلیک روی هر ردیف مشخصات PCR آن نمایش داده میشود.
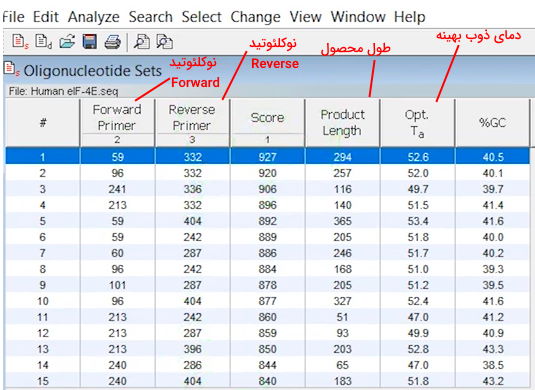
آنالیز پرایمر در نرم افزار Oligo
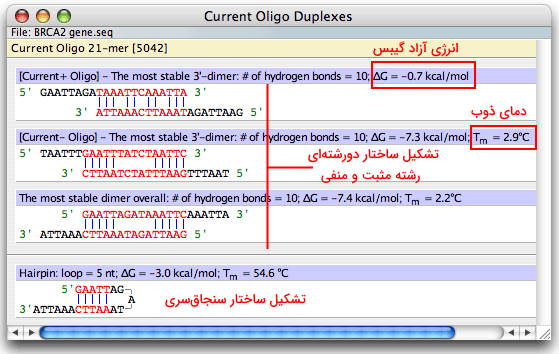
آنالیزها بخش مهمی از آموزش نرم افزار Oligo هستند. از نرمافزار Oligo میتوان برای آنالیز پرایمری که بهوسیله این نرمافزار یا پرایمرهای طراحی شده با روشهای دیگر، استفاده کرد. برای بررسی تشکیل دورشته در توالی اولیگومری یا پرایمر از منوی Analyze آیتم Duplex Formation را انتخاب کنید. پنجره باز شده تشکیل ساختارهای دورشتهای و سنجاقسری را بر اساس دمای ذوب و انرژی آزاد گیبس نشان میدهد. اگر اختلاف دمای ذوب ساختار دوم تشکیل شده با دمای ذوب اولیگومر کمتر از ۱۰ باشد، پرایمر برای تکثیر DNA مناسب نیست.
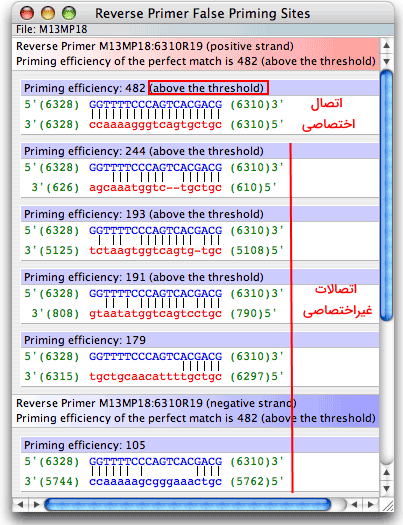
برای اطمینان از تکثیر توالی مورد نظر اتصال اختصاصی پرایمر به DNA باید بررسی شود. این پارامتر را با انتخاب آیتم False Priming Site از منوی Analyze بررسی میکنیم. پنجره این آنالیز تمام توالیهای احتمالی اتصال پرایمرها به رشته مثبت و منفی اولیگومر انتخاب شده را نشان میدهد. دریافت پیام «Above Threshold» نشانه نامناسب بودن پرایمر و احتمال زیاد تشکیل اتصال غیراختصاصی است. تصویر زیر اتصال پرایمر Reverse به رشته مثبت را نشان میدهد. در کادر اول پرایمر به توالی انتخاب شده متصل شده و کادرهای بعدی اتصال غیراختصاصی پرایمر را نشان میدهد. به این نکته توجه کنید که رشته منفی در تمام آنالیزها به رنگ آبی و رشته مثبت قرمز نمایش داده میشود.
«واکنش زنجیره لیگاز» (Ligase Chain Reaction | LCR) یکی دیگر از روشهای تکثیر DNA است که به پرایمر نیاز دارد. در این روش از چهار پروب برای تکثیر DNA استفاده میشود. توالی نوکلئوتیدی این پروبها مکمل توالی تکثیر شونده DNA است. این پروبها در فاصله بسیار نزدیک به رشتههای DNA متصل میشوند. فاصله بین پروبها بهوسیله آنزیم پلیمراز تکمیل میشود و لیگاز مقاوم به گرما دو پروب را به هم وصل میکند. مراحل بعدی این روش شبیه PCR است. به کمک این روش میتوان جهشهای نقطهای را بررسی کرد. برای طراحی پروبهای واکنش زنجیره لیگاز از منوی Analyze آیتم LCR را انتخاب کنید.
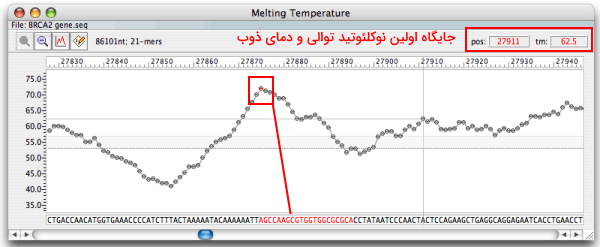
رسم منحنی دمای ذوب
رسم منحنی دمای ذوب یکی دیگر از آنالیزهایی است که نرمافزار Oligo در اختیار کاربر قرار میدهد. برای رسم این نمودار از منوی Analyze آيتم «Melting Temperature Graph» را انتخاب کنید. پنجره باز شده نمودار دمای ذوب تکتک نوکلئوتیدهای توالی انتخاب شده را نشان میدهد. در این پنچره نمیتوانید نوع نمودار را نقطهای یا ستونی انتخاب کنید. هر نقطه یا ستون نشاندهنده توالی ۲۱ نوکلئوتیدی از توالی انتخاب شده در پنجره اصلی است که با کلیک بر روی آن توالی نوکلئوتیدی در نوار پایین پنجره قرمز میشود.
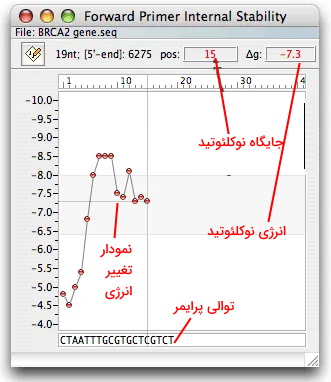
آنالیز پایداری داخلی
پایداری داخلی (انرژی آزاد قند نوکلئوتیدها) یکی دیگر از ویژگیهای مهم پرایمر است. این پارامتر به پیشبینی اختصاصی بودن پرایمر طراحی شده برای PCR و توالییابی را مشخص میکند. برای محاسبه این پارامتر از منوی Analyze آیتم «Internal Stability» و سپس یکی از گزینههای پرایمر یا اولیگومرها را انتخاب کنید. نمودار پنجره باز شده انرژی آزاد گیبس قند هر نوکلئوتید را نشان میدهید. با کلیک بر هر نقطه نمودار جایگاه نوکلئوتید و مقدار انرزی آن در دو کادر سمت راست نمایش داده میشود.
رسم نمودار فراوانی
علاوه بر آنالیزهایی که در بخشهای قبلی آموزش نرم افزار Oligo توضیح دادیم، میتوان به کمک این نرمافزار نمودار فراوانی یک توالی مشخص هگزا یا هپتامری در ژنوم بارگذاری شده را رسم کرد. برای رسم این نمودار از منوی Analyze آيتم «Sequence Frequency» را انتخاب کنید. یا انتخاب هر نقطه روی نمودار نوکلئوتید آن در توالی پایین صفحه قرمز میشود.
آنالیز الگوی خوانش باز
«الگوی خوانش باز» (Open Reading Frame | ORF) بخشی از ژن است که بین کدون شروع و پایان قرار دارد. به کمک منوی Analyze نرمافزار Oligo این نواحی را در یک توالی ژنی مشخص کرد. این آنالیز به انتخاب پرایمر مناسب برای تکثیر DNA یک پروتئین مشخص کمک میکند. با انتخاب گزینه Open Reading Frame پنجره ای باز میشود که در پایین آن توالی نوکلئوتیدها و ترجمه تکحرفی آمینواسید آن و در بخش بالایی طرح شماتیکی از ORF نمایش داده شده است.
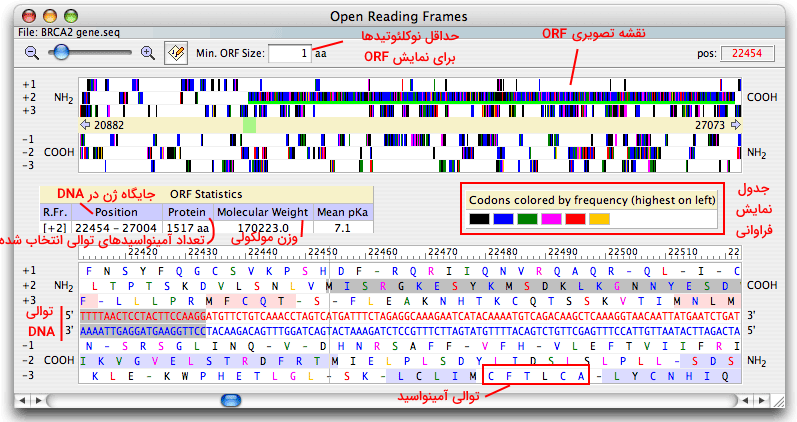
جدول میانی تعداد آمینواسیدها، شماره نوکلئوتیدهای کدکننده ژن، وزن مولکولی و میانگین Pka پروتئین را نشان میدهد. Pka کمیتی است که میزان اسیدی بودن پروتئین در بدن را نشان میدهد. با کلیک روی هر یک از مستطیلهای نوار بالایی مشخصات قطعه پروتئینی در جدول نمایش داده میشود. جدول سمت راست بخش میانی با شش رنگ فراوانی کدونها در ژنوم را نمایش میدهد. رنگ سیاه بیشترین و رنگ زرد کمترین فراوانی را دارد.
آموزش آنالیز آنزیم های محدود کننده
در بسیاری از مطالعات مولکولی و ژنتیکی نیاز است قطع ژنی از سایر بخشهای DNA جدا شود یا با ایجاد برش در زنجیره DNA قطعه اولیگونوکلئوتیدی دیگری به آن اضافه شود. برای انجام این کار از آنزیمهای محدودکننده استفاده میشود. آنزیمهای محدودکننده پروتئینهایی هستند که از باکتریها استخراج میشوند و در توالی مشخصی از DNA برش ایجاد میکنند. بعضی از آنزیمها در هر دو رشته DNA و بعضی در یکی از رشتههای DNA برش ایجاد میکنند. برش بعضی از این آنزیمها انتهای تکرشته و ایجاد میکند و بعضی از آنزیمها دو رشته یکسان برش میدهند. در این بخش از آموزش نرم افزار Oligo روش جست و جوی جایگاه برش آنزیمهای محدودکننده بهوسیله نرمافزار را توضیح میدهیم.
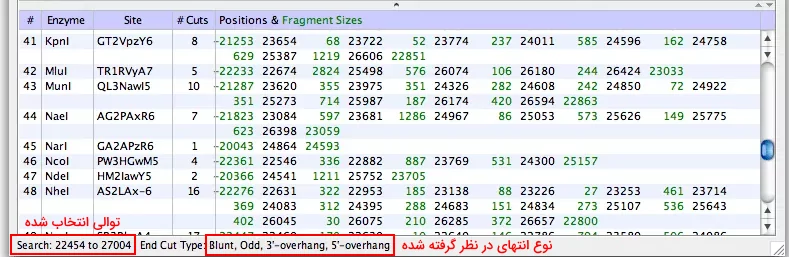
نرمافزار oligo امکان بررسی جایگاه برش آنزیمهای محدودکننده را بر اساس توالی DNA و پروتئین فراهم میکند. این آنالیز در انجام تحقیقات کلونینگ ژن اهمیت ویژهای دارد. برای انجام این آنالیز پس از لود کردن فایل توالی مورد نظر در نرمافزار از منوی Analyze آیتم «Restriction Enzyme Site» را انتخاب کنید. پنجره باز شده نتایج را به دو شکل گرافیکی و آماری نشان میدهد. در بخش گرافیکی سه ستون نمایش داده میشود. ستون اول و دوم شماره آنزیمهای محدودکننده در دیتابیس نرمافزار و در ستون دوم نام آنزیمها نمایش داده میشود. نوار بالایی ستون سوم موقعیت نوکلئوتیدها در ژنوم را نشان میدهد و پایین آن جایگاه برش آنزیم با نوار آبی مشخص شده است.
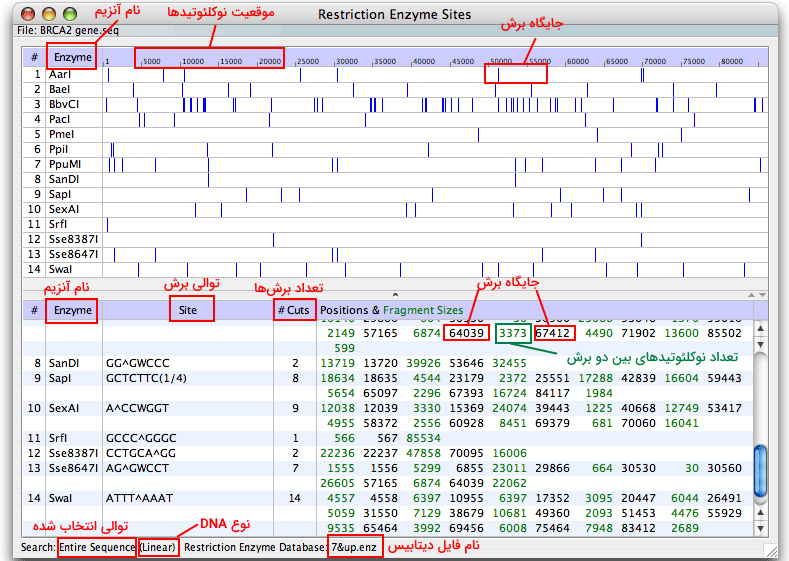
در جدول آماری پنج ستون نمایش داده میشود. ستون اول شماره و ستون دوم نام آنزیم را نشان میدهد. در ستون سوم توالی جایگاه برش مشخص شده و ستون چهارم تعداد برشها بهوسیله آنزیم در کل ژنوم یا توالی انتخاب شده نمایش داده میشود. ستون آخر این جدول جایگاه نوکلئوتید برش با رنگ سیاه و تعداد نوکلئوتیدهای ایجاد شده بین دو نقطه برش با رنگ سبز مشخص میشود. خطی یا حلقوی بدون توالی انتخاب شده، نام قطعه انتخاب شده و نام دیتابیس آنزیمهای محدودکننده نوشته شده است. دیتابیس این نرمافزار شامل ۷۹ آنزیم محدودکننده میشود.
آموزش آنالیز زمان هیبریداسیون
بررسی زمان هیبریداسیون و غلظت بهینه هیبریداسیون آخرین آنالیزی است که در مطلب آموزش نرم افزار Oligo توضیح میدهیم. برای انجام این آنالیز از منوی Analyze آیتم «Hybridization Time» یا «Concentration» را انتخاب کنید. پنجره Hybridization زمان لازم برای اتصال توالیهای اولیگونوکلئوتیدی با اندازه متفاوت به توالی انتهاب شده یا کل ژنوم را در غلظتهای مختلف نشان میدهد. نرمافزار نیمزمان لازم برای هیبریداسیون بهوسیله فرمول زیر محاسبه میکند. در این فرمول N طول و C غلظت اولیگونوکلئوتید است.
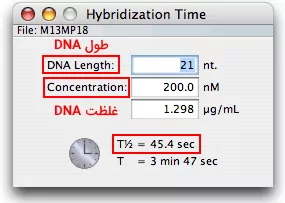
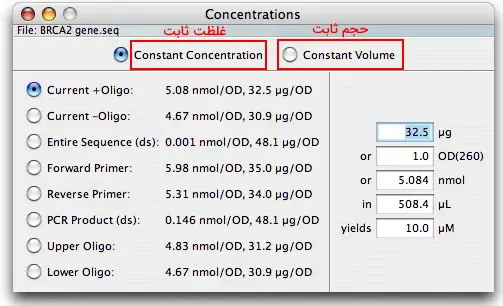
پنجره Concentration غلظت لحظهای، حجم، جذب و وزن مولکولی پرایمرها، محصول PCR و DNA الگو را نشان میدهد. در این پنجره میتوانید غلظت یا حجم را ثابت در نظر بگیرید.
سوالات متداول
در این بخش از مطلب مجله فرادرس به تعدادی از سوالات متداول پیرامون آموزش نرم افزار oligo پاسخ میدهیم.
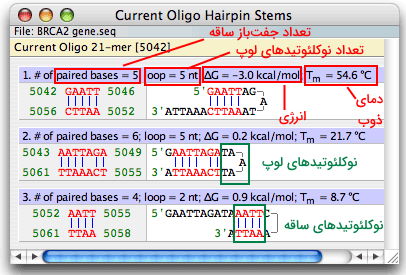
چگونه می توان تشکیل ساختار سنجاق سری در را در نرم افزار oligo بررسی کرد؟
برای بررسی تشکیل ساختار سنجاقسری کاربر میتواند از منوی Analyze گزینه hairpin و سپس یکی از آیتمهای پرایمرهای Forward یا Reverse، توالی انتخابی یا توالیهای بالادست و پایین دست توالی مدنظر را انتخاب کند. پنجره باز شده دمای ذوب، تعداد نوکلئوتیدهای تشکیلدهنده لوپ و تعداد جفتنوکلئوتیدهای ساقه ساختار سنجاقسری را نشان میدهد.
چگونه می توان با استفاده از توالی پروتئینی جایگاه آنزیم های محدودکننده را در نرم افزار oligo بررسی کرد؟
برای بررسی جایگاه آنزیم محدودکننده با استفاده از توالی پروتئينی پس از بارگذاری فایل در نرمافزار از منوی Analyze آیتم «Restriction Enzyme Site in Protein» انتخاب کنید. پنجره باز شده در این آنالیز شبیه پنجره آنالیز آنزیم محدودکننده بر اساس DNA است، با این تفاوت که در نوار پایینی پنجره کنار توالی انتخاب شده، نوع برش آنزیمی نظر گریته شده نمایش داده میشود. "Overhang" نشانه انتهای تکرشتهای، «Blunt» نشانه انتهای دورشتهای و «Odd» نشانه سایر موارد است. در این آنالیز نرمافزار رشته مثبت را بررسی میکند.