طراحی پرایمر چیست؟ – هرآنچه باید بدانید
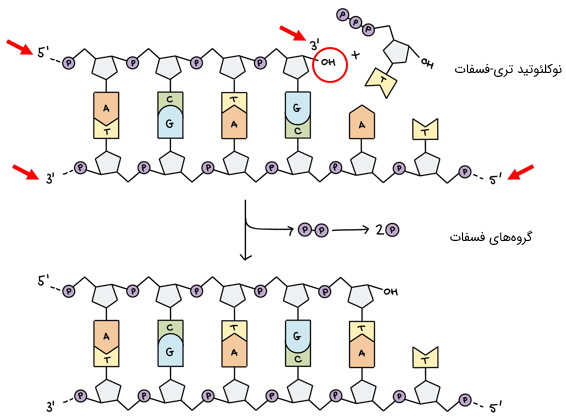
پرایمر، یک مولکول کوچک تک-رشتهای از جنس DNA است که در واکنش PCR شرکت میکند و سر هیدروکسیل آزاد را در اختیار «آنزیم Taq پلیمراز» قرار میدهد. میدانیم که در ژنوم موجودات پیچیدهای مانند انسان، در هر ناحیه، تنها یکی از دو رشته DNA به عنوان ژن فعال، عمل میکند. معمولا هدف از انجام PCR، تکثیر این ناحیه است و به آن «ناحیه هدف» (Target Region) میگوییم. برای این کار به دو پرایمر نیاز است که یکی به رشته هدف و دیگری به رشته مکمل آن متصل میشود.


این دو پرایمر، به ترتیب، «پیشرو» (Forward) و «معکوس» (Reverse) نام دارند و با حروف اختصاری F و R نشان داده میشوند. اتصال پرایمرها به مولکول الگو بر پایه پیوندهای هیدروژنی استوار است و در هر رشته، سر پرایمر به سر مولکول الگو متصل میشود.
اتصال پرایمر
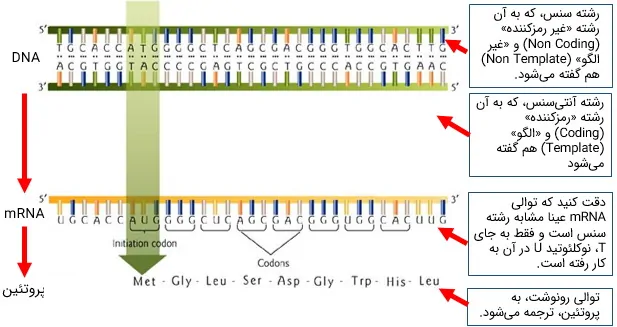
گفتیم در هر ناحیه، تنها یکی از دو رشته DNA حاوی ژن فعال است. این رشته را «آنتیسنس» (Anti-Sense) مینامیم و توالی آن، در واقع، معکوس آن چیزی است که به عنوان توالی ژن، در پایگاههای اطلاعاتی ثبت شده است.
طبق یک قرارداد جهانی، همه پایگاههای اطلاعاتی، توالی رونوشت ژن - و نه خود ژن - را ارائه میکنند. به همین دلیل است که در همه این پایگاهها، کدون آغاز تمامی توالیهای رمزکننده پروتئین، «ATG» است که مستقیما به متیونین ترجمه میشود. این توالی، در واقع همان «AUG» است که در «mRNA» وجود دارد و از روی مکمل خود (یعنی TAC) در DNA آنتیسنس رونویسی شده است.
رشته دیگر DNA که در ناحیه مورد نظر، هیچ توالی رمزکنندهای در آن دیده نمیشود، «سِنس» (Sense) نام دارد و از آن جهت به این نام خوانده میشود که توالی آن از جهت دقیقا مطابق رونوشت (mRNA) است. پرایمر پیشرو، میبایست به رشته آنتیسنس متصل شود. بنابراین به گونهای طراحی میشود که نوکلئوتید انتهای آن مکمل نوکلئوتید مربوط، در انتهای ناحیه هدف در رشته آنتیسنس باشد.
به این ترتیب، اگر بر روی رشتههای پرایمر و رونوشت - که توالی آن به عنوان ژن، پذیرفته شده است - در جهت حرکت کنیم، با ترتیب یکسانی از نوکلئوتیدها برخورد خواهیم کرد. به همین دلیل، این پرایمر را «پیشرو» (Forward) مینامیم. همین قضیه در مورد پرایمر معکوس نیز صادق است. این پرایمر به رشته DNA مقابل (رشته سِنس) متصل میشود و توالی آن از جهت ، عکس و مکمل رشته هدف (ژن) است.
طراحی پرایمر
پیش از آنکه به بررسی اصول طراحی پرایمرها بپردازیم، میبایست با دو اصطلاح علمی آشنا شویم.
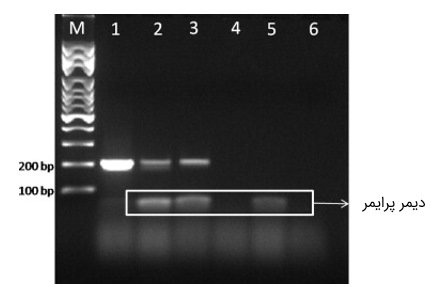
- «کارایی» (Efficiency). این اصطلاح، بیان میکند که پرایمر، تا چه حد در اتصال به توالی مکمل خود در ناحیه هدف و ایجاد محصول نهایی، موفق بوده است. کارایی بالای یک جفت پرایمر، منجر به ایجاد مقدار قابل توجهی از محصول و در نتیجه وجود یک باند بسیار قوی در ژل الکتروفورز خواهد شد.
- «اختصاصیت» (Specificity): این اصطلاح، بیان میکند که آنزیم، تا چه حد، میتواند فقط به توالی مکمل خود در ناحیه هدف، و نه هیچ نقطه دیگری از رشته الگو متصل شود. اختصاصیت بیشتر یک جفت پرایمر، به معنی کاهش باندهای غیراختصاصی در ژل الکتروفورز است.
گاهی حفظ این دو ویژگی در یک جفت پرایمر، با دشواریهایی همراه میشود به طوریکه در بیشتر موارد، افزایش یکی از این دو نیازمند کاهش دیگری است. از سوی دیگر در طراحی پرایمر، میبایست، نکاتی مانند دمای آنیلینگ، درصد GC و مواردی از این دست نیز در نظر گرفته شوند که ترکیب این موارد، فرایند طراحی پرایمر را به یک مبحث پیچیده و در عین حال کلیدی تبدیل میکند.
ویژگیهای موثر بر روی کارایی و اختصاصیت پرایمر عبارتند از:
- طول پرایمر
- «دمای ذوب پرایمر» (Melting Temperature)
- «دمای اتصال پرایمر به الگو» (Primer Annealing Temperature)
- درصد GC در طول توالی پرایمر (GC-Content)
- گیره GC (یا GC Clamp)
- ساختارهای ثانویه (Secondary Structures)
- توالیهای تکراری (Repeats)
- وجود تکرارهای پشت سر هم از یک نوکلئوتید (Run)
- پایداری سر
- جلوگیری از تشکیل ساختار ثانویه در مولکول الگو
طول پرایمر
معمولا اختصاصیت یک پرایمر با تغییر در طول این مولکول، تنظیم میشود که به نوبه خود، منجر به تغییر دمای مرحله آنیلینیگ خواهد شد. در بیشتر موارد، پرایمرهایی به طول 18 تا 24 نوکلئوتید، از اختصاصیت بالایی برخوردارند و در این بازه، افزایش هر نوکلئوتید، موجب تقویت ۴ برابری اختصاصیت پرایمر میشود. کاهش طول این رشتهها به شدت بر توانایی اتصال اختصاصی آنها اثرگذار خواهد بود، به طوریکه پرایمرهایی با طول کمتر از 15 نوکلئوتید، عملا به هر نقطه از ژنوم متصل میشوند.
هرچند این ویژگی، موجب ناکارامدی آنها در PCRهای رایج میشود، اما همین اتصال تصادفی، زمینه را برای مصرف آنها در واکنشهای خاص، مانند «PCR با پرایمرهای دلبخواهی» (Arbitrary PCR) فراهم میکند. این واکنش در تهیه نقشه ژنومی موجودات ساده به کار میرود.
هرچند افزایش طول پرایمرها، محدودیتی ندارد و آنها را میتوان بسیار بلند طراحی کرد، اما باید همواره به این نکته توجه داشته باشید که افزایش طول پرایمر، موجب بیشتر شدن دمای مرحله آنیلینیگ و زمان آن خواهد شد و علاوه بر این، احتمال تشکیل ساختارهای ثانویه و دیمرها را افزایش خواهد داد.
به طور کلی، پرایمرهایی با طول 28 تا 35 نوکلئوتید، تنها زمانی استفاده میشوند که نمونه الگو، سطوحی از «ناهمگنی» (Heterogenicity) را نشان دهد، یا پژوهشگر بخواهد یک دنباله خاص - مانند یک جایگاه برش آنزیمی - را به آن متصل کند. روشن است که در این حالت، بخشی از توالی پرایمر، در دور اول PCR به الگو متصل نخواهد شد. برای حل این مشکل، دمای آنیلینگ را در دورهای اول، پایینتر در نظر میگیرند و سپس، در دورهای بعد، آن را تا حد مجاز افزایش میدهند.
پرایمر
«دمای ذوب» (Melting Temperature | ) یک پرایمر، دمایی است که در آن، نیمی از مولکولهای دورشتهای به حالت تک-رشتهای درمیآیند. این ویژگی، بازتابی از پایداری مولکولهای دورشتهای است. برای پرایمرهایی با طول 20 نوکلئوتید و کمتر، این دما از فرمول زیر به دست میآید:
که در آن، حروف A و T و C و G، معرف تعداد نوکلئوتیدهای حاوی این بازها در رشته پرایمر هستند.
برای تخمین در پرایمرهای بلندتر، از «تئوری ترمودینامیک نزدیکترین همسایه» (Nearest Neighbor Thermodynamic Theory) استفاده میشود که کمی پیچیدهتر از فرمول بالاست و با استفاده از نرمافزارهای طراحی پرایمر، محاسبه میشود. شواهد حاکی از آن است که معمولا، پرایمرهایی با بین 52 تا 58 درجه سانتیگراد، نتایج بهتری را در PCR نشان میدهند.
دمای اتصال پرایمرها
این پارامتر را با نشان میدهند و کاملا تحت تاثیر پرایمرها و محصول قرار دارد. بالا بودن بیش از حد دمای به معنی ناکارامدی هیبریداسیون پرایمر-الگو است که به نوبه خود، منجر به ایجاد یک محصول ضعیف در پایان PCR خواهد شد. از طرفی، پایین بودن بیش از حد این دما نیز، به تولید تعداد زیادی باندهای غیراختصاصی، میانجامد. این باندها در اثر اتصال غیراختصاصی پرایمر به الگو ایجاد شدهاند. دمای از فرمول زیر به دست میآید.
که در آن:
- پرایمر: دمای ذوب ناپایدارترین دوپلکس پرایمر- الگو
- محصول: دمای ذوب محصول PCR
همانطور که از این فرمول پیداست، دمای آنیلینگ، همواره پایینتر از دمای ذوب خواهد بود. در آزمایشگاه، معمولا دمای آنیلینگ، ۲ تا ۴ درجه سانتیگراد، کمتر از دمای ذوب پرایمرها در نظر گرفته میشود.
درصد GC
فراوانی نسبی نوکلئوتیدهای حاوی گوانین (G) و سیتوزین (C) که به صورت درصد بیان میشود، بر روی برخی از ویژگیهای پرایمر، از جمله تاثیرگذار است. معمولا پیشنهاد میشود درصد GC پرایمرها در بازه 40 تا 60 درصد حفظ شود.
گیره GC
وجود نوکلئوتیدهای G و C در بین ۵ نوکلئوتید انتهای پرایمر، با ایجاد پیوندهای شیمیایی پایدارتر بین آن و الگو، به بهبود «کارامدی پرایمر» کمک میکند. به این ترتیب، انتهای پرایمر، مانند یک گیره محکم عمل میکند و آن را به رشته الگو متصل نگه میدارد تا آنزیم پلیمراز، کار ساخت زنجیره جدید را آغاز کند. با این وجود، همواره توصیه میشود تعداد این نوکلئوتیدها در انتهای پرایمر، بیش از ۳ عدد نباشد. چرا که در غیر اینصورت، زمینه را برای ایجاد مشکلاتی مانند تشکیل دیمرهای پایدار در سر فراهم خواهد کرد.
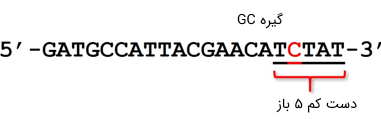
ساختارهای ثانویه پرایمر
ساختارهای ثانویه در اثر برهمکنشهای بین مولکولی و درون مولکولی ایجاد میشوند و یک یا هر دو پرایمر را از دسترس آنزیم پلیمراز خارج میکنند. این مسئله موجب کاهش چشمگیر میزان محصول خواهد شد. این ساختارها در یکی از گروههای زیر قرار میگیرند:
- «سنجاق سرها» (Hairpins): ساختارهای سنجاقسری در نتیجه برهمکنشهای درون-مولکولی شکل میگیرند. در روند طراحی پرایمر، این ساختارها تنها با شرایط زیر نادیده گرفته خواهند شد:
| سنجاق سرهای بخشهای داخلی پرایمر | |
| سنجاق سرهای انتهای پرایمر |
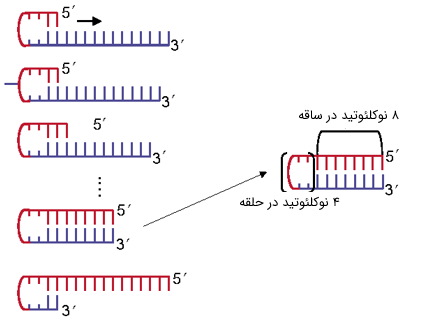
- «خود-دیمر» (Self-Dimer): این ساختارها در اثر برهمکنش بین دو پرایمر از یک نوع رخ میدهند. همانطور که میدانیم، فراوانی پرایمرها در مخلوط واکنش، بسیار بیشتر از ژن هدف است. در این حالت، اگر مثلا پرایمر F طوری طراحی شود که بخشی از توالی آن، مکمل بخش دیگر باشد، در مرحله آنیلینگ، به جای اتصال به مولکول الگو، به پرایمر همتای دیگر متصل خواهد شد و آنزیم نیز، رشته جدیدی را از روی آن سنتز خواهد کرد.
نتیجه این امر، خارج شدن پرایمر از دسترس الگو است که به ضعیف شدن باند محصول در ژل الکتروفورز منجر میشود. در عوض، باندهای کوتاهی در پایین ژل الکتروفورز دیده میشوند که در نتیجه یک تکثیر اشتباهی ایجاد شدهاند. در روند طراحی پرایمر، این ساختارها تنها با شرایط زیر نادیده گرفته خواهند شد:
| دیمرهای خودی در بخشهای داخلی پرایمر | |
| دیمرهای خودی موجود در انتهای پرایمر |

- «دگر-دیمر» (Cross-Dimer): این ساختارها در اثر برهمکنش بین یک پرایمر F و یک پرایمر R شکل میگیرند و پیامدهایی مانند آنچه در مورد خود-دیمرها گفتیم، در پی خواهند داشت. در روند طراحی پرایمر، این ساختارها تنها با شرایط زیر نادیده گرفته خواهند شد:
| دگر-دیمرهای در بخشهای داخلی پرایمر | |
| دگر-دیمرهای موجود در انتهای پرایمر |
توالی های تکراری
منظور از این اصطلاح، وجود یک دی-نوکلئوتید تکراری در طول پرایمر است که مانند توالی «ATATAT» به صورت پشت سرهم تکرار شده است. توالیهایی از این دست، احتمال تشکیل خود-دیمرها را به شدت افزایش میدهند. یک پرایمر، حداکثر مجاز به حمل 4 دی-نوکلئوتید تکراری است.
تکرارها
منظور از این اصطلاح، وجود تکرارهای طولانی از یک نوکلئوتید در طول پرایمر است که ترادفی مانند «ACCCCCCGT» را ایجاد میکنند. چنین تکرارهایی، احتمال اتصال اشتباه پرایمر را افزایش میدهند. یک پرایمر، حداکثر مجاز به حمل 4 نوکلئوتید تکراری پشت سر هم است.
پایداری
این ویژگی، بازتابی از مقدار عددی برای اتصال 5 نوکلئوتید انتهای پرایمر به الگو است. ناپایداری پیوندهای این بخش، منجر به تشکیل باندهای غیر اختصاصی خواهد شد.
ساختارهای ثانویه در مولکول الگو
میدانیم که مولکولهای اسید نوکلئیکی تک رشتهای، بسیار ناپایدارند و با پیچ و تاب خوردن روی خود و تشکیل پیوندهای هیدروژنی بین بخشهای مختلف، انواع ساختارهای ثانویه را ایجاد میکنند. پایداری این ساختارها، به نوبه خود، به «اختلاف میزان انرژی آزاد» () و آنها وابسته است. توجه به مولکول الگو، در طراحی پرایمر انواع واکنشهای PCR، به ویژه «ریل-تایم» (qPCR) از اهمیت بالایی برخوردار است.
اگر هر یک از مولکولهای الگو و پرایمرها ساختارهای ثانویهای را تشکیل دهند که حتی در دماهای بالاتر از دمای آنیلینگ نیز، از هم باز نشوند، این مولکولها قادر به تشکیل پیوند با یکدیگر نخواهند بود. به این ترتیب، میزان محصول به شدت کاهش مییابد و این امر، به ویژه در مورد qPCR - که برای تعیین دقیق و کمی میزان رونوشت یک ژن به کار میرود - موجب خطاهای آشکاری در نتیجه نهایی خواهد شد.
برای حل این مشکل، میبایست، تا حد امکان، پرایمرها برای مناطقی طراحی شوند که احتمال تشکیل ساختارهای ثانویه در آنها ضعیف یا با بالا باشد.
همولوژی (شباهت)
برای بهبود ویژگی «اختصاصیت» پرایمر، باید تا جایی که ممکن است، نقاطی برای اتصال آنها در نظر گرفته شوند که همولوگ بخشهای دیگری از ژنوم یا ترانسکریپتوم نباشند. در غیر اینصورت، پرایمر، علاوه بر ناحیه هدف، در تکثیر نواحی دیگر نیز شرکت خواهد کرد. این پدیده، «اتصال اشتباه» (False Priming) نام دارد و نتیجه آن، ضعیف شدن باند مورد نظر و دیدن باندهای غیراختصاصی در ژل الکتروفورز است. برای حل این مشکل، معمولا از نرمافزار BLAST استفاده میشود. این نرمافزار به صورت رایگان در پایگاه دادههای NCBI موجود است.
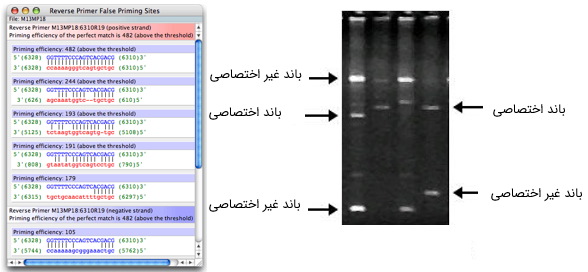
برای شناسایی نواحی یکتا (بدون همولوگ) در ژن مورد نظر، کافیست آن را با ژنوم یا ترانسکریپتوم «غیر تکراری» (non-Redundant) مقایسه کنید. همچنین میتوانید پس از طراحی پرایمرها، توالی هریک از آنها را جداگانه با ژنوم یا ترانسکریپتوم موجود مورد نظر مقایسه کنید. دقت داشته باشید که تشکیل باند اضافی، در صورتی رخ میدهد که دو سوی آن توسط پرایمر، احاطه شده باشد تا پلیمراز بتواند آن را تکثیر کند. برای بررسی میزان همولوژی دو پرایمر F و R، از BLAST توالی این دو مولکول، با هم استفاده کنید.
طراحی یک جفت پرایمر
در این بخش، به معرفی عوامل موثر در طراحی یک جفت پرایمر میپردازیم.
طول محصول
طول محصول PCR، بسته به هدفهای مختلف، متغیر است. معمولا این رقم برای PCR ساده بین 500 تا ۱۰۰۰ جفت باز و برای qPCR حدود 100 جفت باز در نظر گرفته میشود. اگر محل دقیق اتصال هر دو پرایمر مشخص باشد (پرایمرها دلبخواهی و حاوی ترادفهای تصادفی نباشند)، طول محصول PCR از فرمول زیر به دست میآید.
1+ (شماره نوکلئوتید مکمل سر پرایمر F - شماره نوکلئوتید مکمل سر پرایمر R) = طول محصول
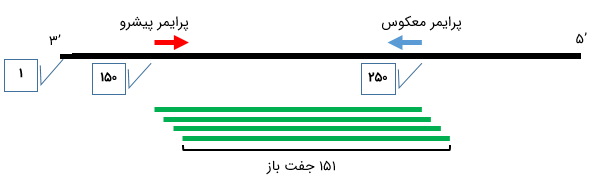
جایگاه محصول
به طور کلی، پرایمرها ممکن است برای هر نقطه دلخواهی از ژن هدف طراحی شوند. اما در واکنشهایی مانند qPCR، نزدیکتر بودن ناحیه تکثیر شده، به سر ژن، نتایج مطمئنتری را به دست میدهد و از این رو بسیار ارجحیت دارد.
محصول
دمای ذوب محصول، در واقع، پایداری دوپلکس پرایمر-الگو را بیان میکند و همانطور که در بالا اشاره شد، از اهمیت بسزایی برخوردار است.
بهینه
فرمولی که در بالا به آن اشاره شد، به فرمول «ریچلیک» (Rychlik) معروف است و در بسیاری از شرکتهای پژوهشی و آزمایشگاههای معتبر، برای محاسبه بهینه به کار میرود.
اختلاف پرایمرها
دمای ذوب دو پرایمر F و R باید یکسان یا دست کم دارای مقدار اندکی تفاوت باشد تا این دو رشته بتوانند، همزمان با هم در مرحله آنیلینگ شرکت کنند و واکنش PCR را به پیش ببرند. بیشینه اختلاف در پرایمرها با هم، ۵ درجه سانتیگراد است.
نکات کلیدی طراحی پرایمر
تا اینجا به بیان اصول کلی طراحی پرایمر پرداختیم. موارد گفته شده را میتوان به صورت ذیل، خلاصه کرد:
- هر دو پرایمر، میبایست های یکسان یا مشابه داشته باشند و این دما، باید ترجیحا از رشته الگو بیشتر باشد (تا در دمایی که رشته الگو هنوز تکرشتهای است، پرایمرها به مکمل خود متصل شوند).
- پرایمرها باید در هیچ یک از دو انتهای یا ، یا دست کم در سر دارای ساختارهای ثانویه نباشند و دیمر تشکیل ندهند.
- نکته: در صورت وجود چنین ساختارهایی، تشکیل آنها میبایست به حدی بالا باشد که در دمای آنیلینگ، مولکول، تمایلی به تشکیل آنها نداشته باشد ().
- نکته: از آنجا که انتهای پرایمر، گروه هیدروکسیل آزاد را در اختیار آنزیم پلیمراز قرار میدهد، در طراحی پرایمر، این بخش، در مقایسه با همواره اهمیت بیشتری دارد.
- نوکلئوتیدها باید به طور کلی، از پراکنش یکسانی در طول پرایمر برخوردار باشند. اما ترجیحا سر غنی از CG (یا GC-Rich) باشد تا اتصالات محکمتری را با الگو برقرار کند.
- ترکیب GC پرایمرها باید با هم و همچنین با محتویاتی که دربرگیرنده مولکول الگو هستند (یعنی ژنوم یا ترانسکریپتوم) تناسب داشته باشد تا در مرحله واسرشتگی، ترکیب نسبتا یکدستی از مولکولهای تک-رشتهای و دورشتهای در مخلوط واکنش وجود داشته باشد.
- پرایمرها، ترجیحا نباید در هیچ بخشی از توالیشان با خود یا پرایمر دیگر، مکمل باشند تا از ایجاد دیمرهای پرایمری جلوگیری شود.
- برای جلوگیری از تشکیل باندهای غیراختصاصی، پرایمرها ترجیحا نباید در هیچ بخشی از توالی خود، با بخشهای دیگری از محتویات دربرگیرنده الگو، مکمل باشند.
- نکته: در صورت وجود چنین بخشهایی، تشکیل آنها میبایست به حدی بالا باشد که در دمای آنیلینگ، مولکول، تمایلی به تشکیل آنها نداشته باشد ().
- احتمال تشکیل باندهای غیراختصاصی و دیمرهای پرایمری را میتوان با انجام BLAST در پایگاه NCBI بررسی کرد.

نرم افزارهای کاربردی طراحی پرایمر
امروزه، نرمافزارهای گوناگونی برای طراحی پرایمر، در دسترس هستند که استفاده از آنها میتواند موجب کاهش چشمگیری در وقت، هزینه و نیروی انسانی شود. نکته قابل توجه در مورد کار با این نرم افزارها، اختلاف در نتیجه خروجی آنها است. این امر، به ویژه برای پژوهشگران کمتجربهتر، ممکن است تا حدی گیجکننده باشد.
توجه داشته باشید که این اختلاف، در بیشتر موارد، ناشی از الگوریتمی است که این برنامهها با آن کار میکنند و گاهی، با تغییر برخی از پیشفرضهای برنامه، به نتایج یکسانی دست خواهید یافت. چند مورد از نرمافزارهای پرکاربرد در زمینه طراحی پرایمر در ادامه آورده شدهاند.
- «Oligo» یکی از پرکابردترین و کاربرپسندترین نرمافزارهای طراحی پرایمر به شمار میرود. این برنامه، به پژوهشگر امکان میدهد تا یک جفت پرایمر یا پروب، یا مجموعهای از آنها را طراحی، آنالیز و با هم مقایسه کند. این آنالیزها شامل بررسی ویژگیهای مختلف پرایمرهای پیشنهادی، از جمله ساختارهای ثانویه، جایگاههای اتصال اشتباهی (False Priming Sites)، پایداریهای داخلی و برخی ویژگیهای فیزیکی است. با اتصال به اینترنت، اولیگو میتواند برخی از دادههای مورد نیاز خود را مستقیما از پایگاه NCBI دریافت و ذخیره کند.
- «Primer Premier» به طور خودکار، کلیه دستورالعملهای لازم برای طراحی یک جفت پرایمر خوب را پیادهسازی میکند. با کمک آن میتوانید برای یک توالی تکی، یا یک مجموعه «همردیفشده» (Alignment) پرایمرهای اختصاصی یا تصادفی (Degenerate) طراحی کنید. این برنامه، همچنین امکان بررسی جایگاههای برش آنزیمی، آنالیز قطعات کانتیگ و طراحی پرایمر برای تعیین توالی را فراهم میکند.
- «AlleleID» و «Beacon Designer» دو نرمافزاری هستند که برای qPCR مناسبسازی شدهاند. این دو برنامه، در طراحی پرایمر و پروبهای مورد نیاز برای شناسایی کمپلکسها به کار میروند.
- «PrimerPlex» برنامهای است که در آن میتوانید پرایمرهای مورد نیاز برای PCR چندگانه (Multiplex PCR) و مطالعات چندگانه SNP در تعیین ژنوتیپ را طراحی کنید.

در این مقاله به معرفی پرایمر و اصول طراحی آن پرداختیم. ویژگیهای یک پرایمر خوب را بررسی کردیم و پس از معرفی دوره آموزشی مفید «طراحی پرایمر با نرم افزار الیگو» با برخی از نرم افزارهای مطرح این حوزه نیز آشنا شدیم. شالوده بحث، این بود که پرایمرها باید طوری طراحی شوند که از طرفی کاملا مکمل توالی هدف باشند و از سوی دیگر به هیچ بخش دیگری از مولکول الگو، متصل نشوند.







باسلام، مطلب خیلی خوب و مفیدی بود. سپاسگزارم
ممنون از توضیحاتتون
ساختار های ثانویه قابل قبول را در ncbi blast چگونه میتوان تشخیص داد ؟
سلام
خیلی عالی و روان توضیح دادید ، بسیار ممنون