معادلات برآوردیابی تعمیم یافته (GEE) در آمار | به زبان ساده
فرض کنید دادههایی به صورت اندازههای تکراری از متغیر پاسخ و همچنین «متغیرهای همبسته» (Covariates Variables) در یک گروه از آزمودنیها داریم. میخواهیم برای متوسط یا میانگین متغیر پاسخ یک مدل مناسب برحسب مشاهدات مجزا و براساس متغیرهای همبسته ایجاد کنیم. توجه داشته باشید که منظور از اندازههای تکراری، استفاده از یک مشاهده در چندین بار آزمایش است که در شرایط مختلف «متغیر گروهبندی» (Category Variables) یا مکان یا زمانهای متفاوت مقدار آن اندازهگیری شده است. این ویژگی باعث ایجاد یک سری داده شده که به آن «دادههای پانلی» (Panel Data) میگوییم. یکی از تکنیکهای بررسی چنین دادههایی استفاده از «معادلات برآوردیابی تعمیم یافته» (GEE) است که خلاصه عبارت (Generalized Estimating Equations) محسوب میشود.
در این نوشتار از مجله فرادرس به کمک زبان محاسبات آماری R، یک مثال را دنبال کرده و موضوعات وابسته به GEE را معرفی میکنیم. به منظور آشنایی بیشتر با دادههای پانلی و تجزیه و تحلیل آنها بهتر است به عنوان مقدمه متن داده پانلی (Panel Data) — از صفر تا صد و آزمون هاسمن برای داده پانلی (Hausman Test) — به زبان ساده را مطالعه کنید. همچنین برای آگاهی از نحوه کار با نرمافزار R و بعضی از توابع آن در سری زمانی، خواندن نوشتارهای مدل هولت وینترز (Holt-Winters) در سری زمانی — راهنمای کاربردی و هموارسازی نمایی سری زمانی — راهنمای کاربردی نیز خالی از لطف نیست.
معادلات برآوردیابی تعمیم یافته (GEE) در آمار
همانطور که در ابتدای متن اشاره شد، به عنوان یک ابزار مدل سازی میتوان از معادلات برآوردیابی تعمیم یافته (GEE) استفاده کرد. زمینههای کاربردی برای GEE میتواند نمونههایی از مسائل زیر باشد.
واضح است که این مسائل وابسته به دادههای پانلی و سریهای زمانی چند بعُدی است.
- نسبت دادن افراد به یک گروه از رژیمهای درمانی و اندازهگیری میزان کلسترول در طول زمان.
- بررسی و تحقیق در مورد رابطه بعضی از متغیر اجتماعی با درآمد افراد در طول زمان.
- تعیین نقش داشتن فرزند برای زنان در احتمال قبولی در مصاحبه شغلی برای نیروی کار.
یکی از مزایای استفاده از دادههای پانلی (سری زمانی یا مکانی با مقادیر تکراری) آن است که میتوانیم روی زمان و مقادیر اختلافات از آزمودنیها (که شاید به صورت یک متغیر پنهان عمل کنند)، کنترل داشته باشیم. از طرفی تکراری بودن مقادیر، باعث میشود برآوردهای حاصل از پراکندگی کمتری برخوردار بوده، در نتیجه برآوردها دقیقتر خواهند بود.
سادهترین روش برای پاسخ به سوالات حوزه مدلسازی، ایجاد یک «مدل خطی» (Linear Model) است که متغیر همبسته در آن با استفاده از یک عملگر جمعی در مدل و روی متغیر وابسته اثر گذار است. در اکثر مواقع با توجه به نوع متغیر وابسته (مثلا مقداری بین صفر و یک یا صحیح یا شمارشی بودن آن) از مدلهای خطی تعمیم یافته استفاده میشود. چنین مدلی را به صورت زیر مینویسند.
$$ \large Y_i = \mu_i + \varepsilon_i, \qquad g(\mu_i) = X_i' \beta $$
در رابطه بالا، $$i$$ نشانگر اندیس مشاهده بوده و $$Y_i$$ نیز مقدار متغیر پاسخ برای فرد $$i$$ام را نشان میدهد. این متغیر میتواند به صورت چند بُعدی و به شکل یک ماتریس باشد. از طرفی $$X_i$$ نیز متغیر همبسته یا کووریت (Covariate) و $$\beta$$ نیز یک بردار از پارامترهای مدل یا ضرایب متغیر مستقل $$X_i$$ است. همچنین $$\epsilon_{i}$$ نیز جمله یا عبارت تصادفی را نشان داده و $$g$$ هم «تابع پیوند» (Link Function) نامیده میشود. تابع $$g$$ یک نگاشت از مجموعه مقادیر ممکن متغیر پاسخ به یک تابع خطی از متغیر $$X$$ است.
به منظور برآورد پارامترهای مدل و استنباط در «مدل خطی تعمیم یافته» (Generalize Linear Model) یا GLM باید فرض کنیم که عبارتهای خطا ($$\epsilon$$) دارای توزیع یکسان (مثلا نرمال) و مستقل از هم (iid) هستند. ولی متاسفانه در دادههای پانلی، چنین امری اتفاق نمیافتد زیرا دادهها به طور تکراری از هر مشاهده اندازهگیری شدهاند و شرط استقلال بین اندازهها برقرار نیست.
یک پاسخ به این مشکل، استفاده از «مدلهای آمیخته خطی تعمیم یافته» (GLMM) است. «مدل آمیخته خطی تعمیم یافته» (Generalize Linear Mixed Model) یک روش پارامتری محسوب میشود و شرط نرمال بودن را برای عبارت خطا، در نظر میگیرد. در عوض «معادلات برآوردیابی تعمیم یافته» (Generalize Estimating Equations) یک روش ناپارامتری بوده و احتیاج به شرط نرمال بودن توزیع عبارت خطا ندارد. از این به بعد برای اشاره به این تکنیک از عبارت GEE که مخفف عبارت لاتین آن است، استفاده خواهیم کرد.
ایده اصلی در GEE، میانگینگیری روی همه مشاهدات و پیدا کردن یک حدس برای ساختار «ماتریس کواریانس» (Covariance Matrix) بین آزمودنیها است. در GEE به جای در نظر گرفتن یک توزیع مشخص برای دادهها، با تکنیکهای محاسبات تکراری و سعی و خطا، بهترین پاسخ برای $$\beta$$ تولید میشود تا بیشترین توصیف برای رابطه بین متغیرهای پاسخ و همبسته را داشته باشد.

تاکید بر آزمودنیها در معادلات برآوردیابی تعمیم یافته
در مدل حاصل از GEE، «اثرات میانگین جامعه» (Population Average Effects) مورد بررسی قرار گرفته و برآورد میشوند. برای درک بهتر این موضوع به دو سناریو زیر توجه کنید.
- سناریو ۱: فرض کنید شما یک پزشک هستید. میخواهید بدانید که مصرف «داروی استاین» (Statin Drug) روی بیماران به چه میزان بخت یا شانس حمله قلبی را کاهش میدهد.
- سناریو ۲: باز هم فرض کنید که شما رییس مرکز بهداشت یک استان هستید. میخواهید بدانید که در صورت استفاده افراد پر خطر از داروی استاین، به چه میزان مرگ و میر ناشی از سکته قلبی در جامعه کاهش یا تغییر خواهد یافت.
در سناریو اول، متغیر پاسخ شانس یا بخت است و در سناریو دوم، متغیر پاسخ تعداد است. همچنین در سناریو اول، در مورد افراد بیمار صحبت میکنیم ولی در سناریو دوم، در مورد کل جامعه قضاوت و بررسی صورت خواهد گرفت. توجه داشته باشید که استفاده از GEE میتواند در سناریو دوم به کار آید و برای تحلیل سناریو اول کاربرد نخواهد داشت.
کالبد شکافی معادلات برآوردیابی تعمیم یافته
همانطور که گفته شد، GEE پارامترهای مدل میانگین جمعیت و خطاهای استاندارد آنها را تخمین میزند. فرضیات مربوط به GEE مشابه فرضیههای GLM است. به بعضی از این پیشفرضها در ادامه اشاره خواهیم کرد.
- مقادیر متغیر پاسخ که به صورت $$Y_1, Y_2 , \ldots , Y_n$$ مشخص میشوند، با یکدیگر ارتباط داشته یا تشکیل «خوشهای» (Cluster) میدهند.
- بین متغیرهای $$X$$ یا همان کووریتها و تغییر پاسخ، که توسط تابع پیوند $$g$$ توصیف شده است، یک رابطه خطی وجود دارد.
- در GEE باید ماتریس کوواریانس درون آزمودنیها را براساس هر یک از ساختارهای زیر در نظر گرفت.
- با حفظ «شرط استقلال» (Independence Working Covariance) که در این حالت مشاهدات نسبت به گذشت زمان یا طی شدن هفتهها، مستقل هستند.
- قابلیت «تعویضپذیری برای ماتریس کوواریانس» (Exchangeable Working Covariance) که در این صورت تمام مشاهدات در طول زمان با هم ارتباط دارند.
- به کارگیری «ماتریس کوواریانس به صورت خودهمبسته مرتبه اول» (AR (1 یا Autoregressive که هر مشاهده با مشاهده دیگر برحسب تعداد هفتههای سپری شده، مرتبط است. برای مثال اگر مدل (AR(1 در نظر گرفته شده، فاصله زمانی بین مشاهدات یک هفته محسوب خواهد شد.
- مدل GEE بدون در نظر گرفتن هیچ ساختاری برای ماتریس کوواریانس که به این ترتیب ارتباط یا همبستگی بین تمامی زمانها برای مشاهدات، ممکن است در ماتریس کوواریانس متفاوت باشد.
در ادامه سه مورد اول را با توجه به ساختار و مدلهای ماتریس کوواریانس مطرح کرده و برای ایجاد مدل GEE به کار خواهیم برد. همانطور که گفته شد در تکنیک GEE یک روش تکراری به منظور بهبود مدل با استفاده از «کمترین مربعات وزندار» (Iteratively Reweighted Least Squares) انجام میشود که در آن وزنها براساس ماتریس کوواریانس مشخص میشوند.
مسئلههای کمترین مربعات وزندار در حقیقت همان معادلات برآوردیابی هستند. اگر نسبت به تکنیک «بیشترین درستنمایی» (Maximum Likelihood) آگاهی داشته باشید، میتوانید «تابع امتیاز» (Score Function) را در آنجا، مشابه «معادلات برآوریابی» (Estimating Equation) در نظر بگیرید. مقدار این معادلات در زمانی که بهترین پاسخ برای $$\beta$$ حاصل شود، برابر با صفر است.
باید توجه داشت که تکنیک GEE یک روش «نیمهپارامتری» (Semi-Parametric) است. هر چند ما، برخی ساختارها را بر فرآیند تولید مدل (مثل خطی بودن) در نظر گرفتهایم، اما توزیع آن را به طور کامل تعیین نکردهایم. مشخص است که در اینجا بدست آوردن بردار $$\beta$$ یک «مسئله بهینهسازی» (Optimization Problem) است.
مشخص کردن ماتریس کوواریانس در معادلات برآوردیابی تعمیم یافته
هنگامی که بخواهیم یک مدل GEE ایجاد کنیم باید در مورد نحوه انتخاب ساختار ماتریس کوواریانس به بررسی بپردازیم. ممکن است در این هنگام سوالاتی نظیر سوالات زیر به ذهنتان برسد.
- نگران این هستم که ماتریس کوواریانس به درستی مشخص نشده باشد، در این صورت چه باید بکنم؟
- برای برآورد ضریبهای مدل ($$\beta$$) باید ساختار کوواریانس را انتخاب کنیم، اما اگر این ساختار درست نباشد چقدر میتوان به نتایج اعتماد کرد؟
از آنجا که معادلات تخمین زده شده بر اساس اولین گشتاور (میانگین) ساخته میشوند، ضرایب $$\beta$$، برآوردگرهای سازگار (Consistence Estimators) هستند، حتی اگر ساختار ماتریس کوواریانس به کار رفته، اشتباه باشد. با این حال، «خطاهای استاندارد» (Standard Error) محاسبه شده در این حالت، اشتباه یا بسیار بزرگ خواهد بود.
برای رفع این مشکل و ایجاد یک مدل GEE «استوار» (Robust)، از «برآوردگرهای فشرده» (Sandwich Estimator) که به برآوردگرهای «هوبر-وایت» (Huber-White) نیز معروف هستند، استفاده میکنیم. ایده اصلی در برآوردگر واریانس فشرده، استفاده از باقیماندههای تجربی برای تقریب ماتریس کوواریانس اصلی است.
به این موضوع نیز توجه داشته باشید که آغاز کار برای ایجاد مدل GEE، مشخص کردن ساختار و مقادیر ماتریس کوواریانس است. این کار به بالا بردن کارایی آماری و به کارگیری صحیح از مدل کمک میکند. از طرفی به یاد داشته باشید که برآوردگر استوار ماتریس کوواریانس براساس نمونه بزرگ صورت میگیرد، بنابراین به کارگیری آن باید با احتیاط صورت گیرد.
معمولا در صورتی که یکی از شرایط زیر در مسئله وجود داشته باشد، نمیتوان از برآوردگر ماتریس کوواریانس استوار یا برآوردگر فشرده آن استفاده کرد.
- تعداد آزمودنیهای مستقل بسیار کمتر از تعداد اندازههای تکراری باشند.
- طرح آزمایش نامتوازن باشد، به این معنی که تعداد اندازههای تکراری در بین آزمودنیها یکسان در نظر گرفته نشده باشد.
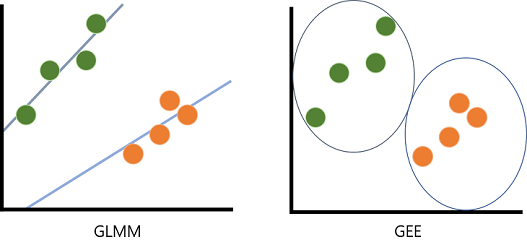
در تصویر بالا، تفاوت بین مدل خطی تعمیم یافته (GLMM) و معادلات برآوردیابی تعمیم یافته (GEE) را میبینید. اولی سعی در ایجاد یک مدل خطی دارد، در حالیکه دومی میخواهد خوشههایی ایجاد کند که به واسطه آن ساختار درونی دادهها استخراج شوند. در ادامه خروجیهای حاصل از GEE را مشاهده میکنید که علاوه بر معرفی پارامترهای مدل، «تعداد خوشهها» (Clusters) و اندازه یا «تعداد اعضای هر خوشه» (Cluster Size) را هم مشخص کرده است.
بررسی یک مثال واقعی برای معادلات برآوردیابی تعمیم یافته
در این قسمت به یک مثال خواهیم پرداخت که از نمونههای معروف برای مدلهای خطی و بخصوص معادلات برآوردیابی تعمیم یافته یا GEE است. یک نمونه از خوکها، تحت رژیمهای غذایی مختلف قرار گرفتهاند و میزان افزایش وزن آنها مورد بررسی قرار گرفته است. سوالی که در اینجا مطرح است میزان اثر ویتامین E و ماده معدنی مس در خوراک آنها، بر وزن خوکها است. در حقیقت میخواهیم بدانیم که سطوح مختلف این تیمارها یعنی دوز ویتامین E در سطوح ۰، ۱۰۰ و ۲۰۰ میلیگرم و ماده معدنی مس (Cu) در سطوح ۰، ۲۵ و ۱۷۵ میلیگرم در هر کیلو غذا به چه شکلی روی افزایش وزن آنها تاثیر گذار است.
دادههای این مسئله به صورت زیر معرفی میشوند.
- اندازه وزن خوکها در پایان هر هفته. این کار در طول ۱۲ هفته کامل (حدود سه ماه) تکرار شده است.
- وزن در آغاز طرح (یعنی وزن در هفته اول).
- مصرف خوراک به صورت تجمعی. به این معنی که در آخر هر هفته، کل غذای مصرفی تا آن هفته مشخص شده است.
تیمارهای صورت گرفته روی خوکها نیز به صورت زیر بوده و از یک طراحی فاکتوریل 3x3 استفاده شده است.
- ویتامین E (دوز: 0 ، 100 ، 200 میلی گرم در هر کیلوگرم خوراک)
- مس (دوز: 0 ، 35 ، 175 میلی گرم در هر کیلوگرم خوراک)
به منظور پیادهسازی مدل GEE از کتابخانه geepack استفاده کرده و چند مدل یا حالت را برای ماتریس کوواریانس در نظر میگیریم. ابتدا بارگذاری کتابخانه و آماده سازی دادهها را اجرا میکنیم، سپس مدلهای مختلف GEE برحسب ماتریسهای کوواریانس متفاوت را روی این دادهها به کار میگیریم.
1library("geepack")
2data(dietox)
3dietox$Cu <- as.factor(dietox$Cu)
4dietox$Evit <- as.factor(dietox$Evit)
5mf <- formula(Weight ~ Time + Evit + Cu)
6head(dietox)
7 <div class="faradars-courses faradars-courses-single">
8 <a href="https://faradars.org/how-to-learn/r-programming-and-rstudio?utm_source=blog.faradars&utm_medium=referral-post&utm_campaign=related-courses-inline-5&utm_term=a.reybod&utm_content=statistics" target="_blank">
9 <div class="card card-course">
10 <div class="card-image">
11 <img class="pop-img" src="https://faradars.org/wp-content/uploads/2019/03/r-3-1.svg" alt="مجموعه آموزش برنامه نویسی آر R و RStudio – مقدماتی تا پیشرفته">
12 </div>
13 <div class="card-body">
14 <div class="card-title">
15 فیلم مجموعه آموزش برنامه نویسی آر R و RStudio – مقدماتی تا پیشرفته در فرادرس
16 </div>
17 </div>
18 <div class="card-action ml-3">
19 <div class="fdb-btn">کلیک کنید</div>
20 </div>
21 </div>
22 </a>
23 </div>
24 سطر اول کتابخانه geepack برای استفاده از مجموعه داده را بارگذاری کرده و توابع مورد نظر را در حافظه قرار میدهد. همچنین تبدیل متغیرهای Cu و Evit به «متغیرهای عامل» (Factor) در سطرهای بعدی دیده میشوند. مدل ارتباط بین متغیر وابسته یا پاسخ (Weight) با متغیرهای همبسته یعنی زمان (Time) و دوز ویتامین (Evit) و مس (Cu)، به صورت خطی و جمعی است. سطر آخر نیز به نمایش این دادهها پرداخته است.
پس از اجرای این برنامه، نتایج محاسبه شده طبق خروجی زیر خواهد بود.
1## Weight Feed Time Pig Evit Cu Litter
2## 1 26.50000 NA 1 4601 1 1 1
3## 2 27.59999 5.200005 2 4601 1 1 1
4## 3 36.50000 17.600000 3 4601 1 1 1
5## 4 40.29999 28.500000 4 4601 1 1 1
6## 5 49.09998 45.200001 5 4601 1 1 1
7## 6 55.39999 56.900002 6 4601 1 1 1البته به کمک دستور (View(dietox میتوانید یک نمای کلی از دادهها، مشاهده کنید. در تصویر زیر چند سطر اول از این مجموعه داده قابل مشاهده است. همانطور که میبینید، هر یک از خوکها ۱۲ بار اندازهگیری شده و سطرهای تکراری را تشکیل دادهاند. شماره مشخصه مربوط به هر خوک در ستون pig دیده میشود.
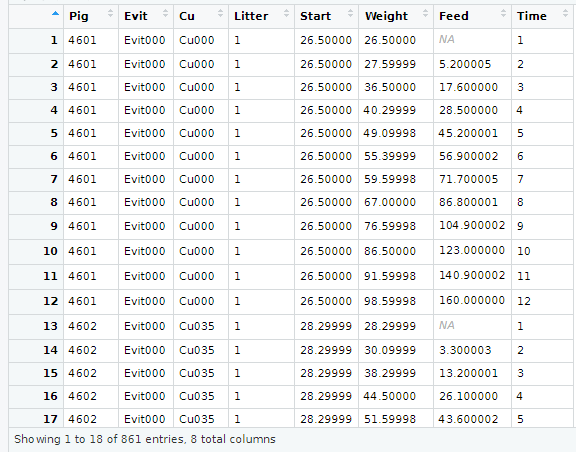
متغیرهای مورد نظر در اینجا به ترتیب، ستون Pig شماره آزمودنی، ستون Evit، دوز یا طبقه مربوط به میزان ویتامین E، ستون Cu سطح دوز ماده معدنی مس، ستون Litter، محل نگهداری حیوان، ستون Start، وزن در آغاز اجرای طرح، ستون Weight، وزن در هر هفته و ستون Feed که میزان خوراک را به صورت جمعی برای هر حیوان نشان میدهد، به این معنی که در این ۱۲ هفته، این ستون، میزان خوراک مصرفی در پایان هر هفته مشخص شده است. در ستون TIme نیز هفته مربوط به اندازهگیری تعیین و نمایش داده شده است.
واضح است که وزن 72 خوک در ۱۲ نوبت ماهانه، مورد بررسی قرار گرفته است. البته برای سه مورد در ماه ۱۲، اندازهگیری صورت نگرفته است. بنابراین این طرح «نامتوازن» (Unbalanced) است به این معنی که تعداد اندازههای تکراری در هر یک از آزمودنیها، متفاوت است.
با توجه به تصویر بالا، اندازههای تکراری برای هر آزمودنی، به خوبی مشخص میشوند. البته از همه این متغیرها (مثلا متغیر Litter یا Start) استفاده نخواهد شد و در مدل خطی نقشی نخواهند داشت. چارچوب دادهای به نام mf، اصل کار ما را تشکیل داده و از بقیه متغیرها ساخته شده است. همچنین این چارچوب داده (DataFrame)، ساختار متغیرهای پاسخ و همبسته را تعیین کرده است.
نکته: توجه داشته باشید که این مجموعه داده، درون کتابخانه geepack قرار دارد و لازم نیست آن را از خارج از برنامه R، فراخوانی یا بارگذاری کنید.
مدل GEE با فرض استقلال در ماتریس کوواریانس
در مدلی که کد زیر تولید میکند، فرض مربوط به استقلال بین مشاهدات و در نتیجه ماتریس کوواریانس برقرار است. برای این کار از تابع geeglm استفاده شده است.
1geeInd <- geeglm(mf, id=Pig, data=dietox, family=gaussian, corstr="ind")
2summary(geeInd)خانواده توزیع برای مدل GEE در اینجا توزیع گاوسی در نظر گرفته شده است. ساختار ماتریس کوواریانس با پارامتر "crostr="ind، به شکل مستقل فرض شده است. نتیجه محاسبه منجر به تولید خروجی زیر خواهد شد.
1##
2## Call:
3## geeglm(formula = mf, family = gaussian, data = dietox, id = Pig,
4## corstr = "ind")
5##
6## Coefficients:
7## Estimate Std.err Wald Pr(>|W|)
8## (Intercept) 15.07283 1.42190 112.371 <2e-16 ***
9## Time 6.94829 0.07979 7582.549 <2e-16 ***
10## Evit2 2.08126 1.84178 1.277 0.258
11## Evit3 -1.11327 1.84830 0.363 0.547
12## Cu2 -0.78865 1.53486 0.264 0.607
13## Cu3 1.77672 1.82134 0.952 0.329
14## ---
15## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
16##
17## Estimated Scale Parameters:
18## Estimate Std.err
19## (Intercept) 48.28 9.309
20##
21## Correlation: Structure = independenceNumber of clusters: 72 Maximum cluster size: 12آنچه مدل را ساخته است، «عرض از مبدا» (Intercept) و متغیر «زمان» (Time) است و دیگر عوامل، در مدل دارای ضرایب معنیداری نیستند. توجه داشته باشید که سطح اول برای «متغیر ویتامین» (Evit) و «مس» (Cu) که نشانگر عدم استفاده از این افزونههای غذایی است، در عرض از مبدا یا همان مقدار ثابت مدل، نقش داشته و سطوح بعدی معنیدار نیستند. سطحهای دیگر این متغیرها (که میزان دوزی مخالف صفر دارند) به صورت Evit2, Evit3 و Cu2, Cu3 مشخص شدهاند.
حال به سراغ تحلیل واریانس مدل حاصل میرویم. از کد زیر به این منظور استفاده کردهایم.
1anova(geeInd)جدول آنالیز واریانس به صورت زیر در خواهد آمد.
1## Analysis of 'Wald statistic' Table
2## Model: gaussian, link: identity
3## Response: Weight
4## Terms added sequentially (first to last)
5##
6## Df X2 P(>|Chi|)
7## Time 1 7507 <2e-16 ***
8## Evit 2 4 0.15
9## Cu 2 2 0.41
10## ---
11## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1همانطور که دیده میشود، فقط متغیر زمان در افزایش وزن از لحاظ آماری و در سطح خطای ۰٫۰۵، معنیدار و اثر گذار است و بقیه متغیرهای عامل، با اهمیت تلقی نمیشوند.
مدل با کوواریانس تعویضپذیر
فرض کنید که ماتریس کوواریانس به صورت «قابل تعویض» (Exchangeable) در معادلات برآوردیابی تعمیم یافته به کار رفته باشد، یعنی تقارن در آن وجود داشته یا همه وابستگیها یکسان هستند. در این صورت دستور زیر مدل را ایجاد خواهد کرد. واضح است پارامتر corstr برابر با ex قرار گرفته.
1geeEx <- geeglm(mf, id=Pig, data=dietox, family=gaussian, corstr="ex")
2summary(geeEx)خروجی تابع geelm به صورت زیر ظاهر میشود. توجه دارید که نتایج در یک کلاس به نام geeEX از نوع GEE ذخیره شده است.
1##
2## Call:
3## geeglm(formula = mf, family = gaussian, data = dietox, id = Pig,
4## corstr = "ex")
5##
6## Coefficients:
7## Estimate Std.err Wald Pr(>|W|)
8## (Intercept) 15.0984 1.4206 112.96 <2e-16 ***
9## Time 6.9426 0.0796 7605.79 <2e-16 ***
10## Evit2 2.0414 1.8431 1.23 0.27
11## Evit3 -1.1103 1.8452 0.36 0.55
12## Cu2 -0.7652 1.5354 0.25 0.62
13## Cu3 1.7871 1.8189 0.97 0.33
14## ---
15## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
16##
17## Estimated Scale Parameters:
18## Estimate Std.err
19## (Intercept) 48.3 9.31
20##
21## Correlation: Structure = exchangeable Link = identity
22##
23## Estimated Correlation Parameters:
24## Estimate Std.err
25## alpha 0.766 0.0326
26## Number of clusters: 72 Maximum cluster size: 12نتایج حاصل از مدل، درست به مانند قبل است با این تفاوت که ضریب متغیر زمان و عرض از مبدا با مدل قبلی، کمی اختلاف دارند. این بار برای نمایش جدول آنالیز واریانس از دستور زیر استفاده میکنیم.
1anova(geeEx)در ادامه خروجی این دستور را مشاهده میکنید.
1## Analysis of 'Wald statistic' Table
2## Model: gaussian, link: identity
3## Response: Weight
4## Terms added sequentially (first to last)
5##
6## Df X2 P(>|Chi|)
7## Time 1 7604 <2e-16 ***
8## Evit 2 4 0.16
9## Cu 2 2 0.41
10## ---
11## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1در اینجا هم اثرات اصلی تیمارها (مصرف ویتامین و ماده معدنی مس) در سطح خطای ۰٫۰۵، معنیدار نخواهند بود، زیرا برای هر یک از آنها مقدار احتمال در ستون (|P(>|Chi، بزرگتر از ۰٫۰۵ است.
مدل GEE با در نظر گرفتن (AR(1
در مدل زیر فرض بر این است که مشاهدات به صورت «خودهمبسته» (Autoregressive) از مرتبه اول هستند.
1geeAr1 <- geeglm(mf, id=Pig, data=dietox, family=gaussian, corstr="ar1")
2summary(geeAr1)به این ترتیب محاسبات مربوط به برآورد پارامترهای مدل صورت گرفته و خروجی به صورت زیر در خواهد آمد.
1##
2## Call:
3## geeglm(formula = mf, family = gaussian, data = dietox, id = Pig,
4## corstr = "ar1")
5##
6## Coefficients:
7## Estimate Std.err Wald Pr(>|W|)
8## (Intercept) 17.6124 1.3354 173.95 <2e-16 ***
9## Time 6.7324 0.0756 7921.11 <2e-16 ***
10## Evit2 2.3782 1.7676 1.81 0.18
11## Evit3 -0.9779 1.7369 0.32 0.57
12## Cu2 -0.3976 1.3928 0.08 0.78
13## Cu3 1.2376 1.7376 0.51 0.48
14## ---
15## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
16##
17## Estimated Scale Parameters:
18## Estimate Std.err
19## (Intercept) 50.5 9.41
20##
21## Correlation: Structure = ar1 Link = identity
22##
23## Estimated Correlation Parameters:
24## Estimate Std.err
25## alpha 0.933 0.0116
26## Number of clusters: 72 Maximum cluster size: 12همانطور که در مدل معادلات برآوردیابی تعمیم یافته مشاهده میکنید، عرض از مبدا و زمان، در مدل با توجه به اندازه آزمون ۰٫۰۰۱ معنیدار شده است. همچنین اثرات ویتامین E یا (Evit000) و مس (Cu000) در سطح اول در عرض از مبدا لحاظ شدهاند.
درست به مانند قبل، برای نمایش «آماره والد» (Wald Statistics) و مقدار احتمال (p-value) از دستور زیر استفاده کنید.
1anova(geeAr1)به این ترتیب خروجی «تحلیل واریانس» (ANOVA) به صورت زیر در خواهد آمد. مشخص است که در اینجا مقدار احتمال در ستون (|P(>|Chi مشخص شده است.
1## Analysis of 'Wald statistic' Table
2## Model: gaussian, link: identity
3## Response: Weight
4## Terms added sequentially (first to last)
5##
6## Df X2 P(>|Chi|)
7## Time 1 7907 <2e-16 ***
8## Evit 2 5 0.07 .
9## Cu 2 1 0.65
10## ---
11## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1در این حالت، میزان دوزهای مصرف ویتامین E (متغیر Evit) و زمان (Time) برجسب هفته، در افزایش وزن خوکها، معنیدار تشخیص داده شده و مقدار احتمالی کوچکتر از ۰٫1 دارند. این متغیرها در جدول تحلیل واریانس با علامت *** و . مشخص شدهاند. اگر سطح آزمون را برابر با ۰٫۰۵ در نظر میگرفتیم، فقط زمان در تغییر وزن معنیدار محسوب میشد.
نکته: توجه داشته باشید که در همه خروجیهای حاصل از GEE، ۷۲ خوشه با تعداد عضو ۱۲ ایجاد شده است. بنابراین هر خوک در یک خوشه قرار دارد.
مزایا و محدودیتهای استفاده از مدل GEE
یکی از مزایای مهم در به کارگیری مدل GEE نسبت به مدل GLM که برآوردهای آن توسط «حداکثر درستنمایی» (MLE) حاصل میشود، سادگی و راحتی انجام محاسبات است. از طرفی نبودن فرض مشخصی در مورد توزیع عبارت خطا، از دیگر مزیتهای این تکنیک آمار ناپارامتری محسوب میشود. برآوردها حاصل از مدل GEE، «سازگار» (Consentient) هستند. سازگاری حتی زمانی که ساختار همبستگی نادرستی تعیین شده باشد نیز برقرار است به شرط آن که مدل میانگین متغیر پاسخ به شکل درست انتخاب شده باشد.
از طرفی محدودیتهایی نیز برای مدل GEE وجود دارد. روشهای مبتنی بر احتمال و تابع درستنمایی برای استنباط آماری در این حالت قابل استفاده نیست زیرا شرط استقلال یا داشتن توزیع توام با مشکل همراه است. به همین دلیل مشخص است که GEE یک روش «شبه-درستنمایی» (Quasi-likelihood) خواهد بود. همچنین در مورد نحوه انتخاب مدل، تکنیک GEE راهکاری ارائه نمیدهد بلکه فقط روشی برای برآورد محسوب میشود. واضح است که برای «سنجش نیکویی برازش» (Goodness of Fit) هیچ معیاری توسط مدل GEE معرفی نشده است.
نکته: توجه داشته باشید که GEE یک «تحلیل درون آزمودنی» (Within Subject Analysis) نیست و نباید از آن برای مدل سازی روی چنین مسائلی استفاده کرد.
نکتههایی برای به کارگیری معادلات برآوردیابی تعمیم یافته
هنگامی که تحلیل GEE را به کار میبرید، باید به بعضی از شرایط و وضعیتهای مربوط به مسئله و برآوردها حساس باشید. در ادامه لیستی از نکات مهم در هنگام به کارگیری معادلات برآوردیابی تعمیم یافته را متذکر میشویم.
- در GEE اولین شرط وجود «میانگین» (Mean) و «ماتریس کوواریانس» (Covariance Matrix) است. این از الزامات اولیه برای به کارگیری روش «شبه درستنمایی» (Quasi-Lieklihoood) محسوب میشود. به این ترتیب «گشتاور» (Moment) اول و دوم برای توزیع باید موجود باشد.
- برای جلوگیری از سوء تعبیر از ماتریس کوواریانس از «برآوردگرهای فشرده» (Sandwich Estimator) استفاده کنید. برآوردگرهای فشرده نسبت به برآوردگرهای عادی، واریانس کوچکتری دارند ولی ممکن است خاصیت نااریبی نداشته باشند.
- توجه داشته باشید که معادلات برآوردیابی تعمیم یافته اثرات میانگین جمعیت را مدلبندی میکند.
- مدل GEE، زمانی که وابستگی درون آزمودنیها مشاهده نشده یا ناشناخته است، مفید خواهد بود.
- در معادلات برآوردیابی تعمیم یافته باید هر آزمودنی را مستقل از دیگران در نظر بگیرید. البته مشخص است که مشاهدات مربوط به یک آزمودنی با هم همبستگی دارند ولی به شرط متغیر همبسته (Covariate)، آزمودنیها مستقل از یکدیگر خواهند بود.
خلاصه و جمعبندی
در این نوشتار با مفاهیم و روشهای به کار رفته در معادلات برآوردیابی تعمیم یافته یا به اختصار GEE آشنا شدیم. در این بین برای پیادهسازی محاسبات GEE از زبان برنامهنویسی و محاسبات آماری R و کتابخانه geepack کمک گرفتیم. همچنین به کمک مثالی از دادههای واقعی جنبههای مختلف مدل GEE را بررسی و خروجیهای حاصل را نمایش دادیم. همانطور که در مثال دیده شد، شکل ماتریس کوواریانس برای مدل GEE مهم است. در انتهای این متن نیز به مزایا و معایب استفاده از مدل GEE پرداختیم.